CICLO CELULAR Y DUPLICACIÓN DEL ADN
Ciclo celular
Las células de los distintos organismos pasan durante su vida por distintos períodos, cada uno de ellos característico y claramente diferenciado.
Cada tipo celular cumple con sus funciones específicas durante la mayor parte de su vida, creciendo gracias a la asimilación de materiales provenientes de su ambiente y con ellos sintetiza nuevas moléculas por medio de complejos procesos regulados por su material genético.
Cuando una célula aumenta hasta llegar a un determinado tamaño, su eficiencia metabólica se torna crítica, entonces se divide. En los organismos pluricelulares, se produce un crecimiento a partir de una célula (huevo o cigoto) como así también se aumenta la masa tisular y se reparan los tejidos lesionados o desgastados, por aumento del número de células.
Las nuevas células originadas en esta división poseen una estructura y función similares a las células progenitoras, o bien derivadas de ellas.
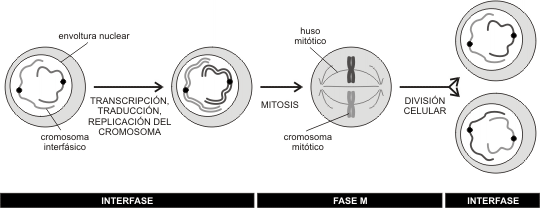
En parte son similares porque cada célula nueva, recibe aproximadamente la mitad de organoides y citoplasma de la célula madre, pero en términos de capacidades estructurales y funcionales lo importante es que cada célula hija, reciba una réplica exacta del material genético de la célula madre.
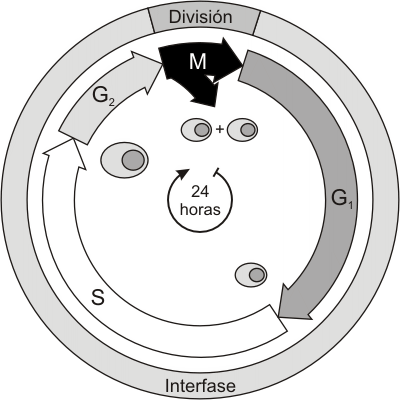
Durante la vida celular, las células pasan por un ciclo regular de crecimiento y división. A esta secuencia de fases se la denomina ciclo celular y en general consta de un período donde ocurre un importante crecimiento y aumento de la cantidad de organoides (interfase) y un período de división celular (mitosis o meiosis).
La interfase involucra períodos donde la célula realiza los procesos vitales propios de su función. Durante ella, se producen también fenómenos a nivel nuclear imprescindibles para la división posterior. Cronológicamente podemos dividir la interfase en tres etapas G1, S y G2.
Haciendo un esquema del ciclo celular, el tiempo en que transcurre cada una de las etapas se representa en la Fig. 12.2.
Es necesario señalar que existen excepciones a este ciclo, ya que no en todas las células los períodos tienen la misma duración. Incluso si consideramos una población celular homogénea (células del mismo tipo), existen variaciones particulares. Siempre que se habla de tiempos determinados, se hace considerando los promedios de cada tipo celular.
También existen células que dejan de dividirse por largos períodos o bien permanentemente. Por ejemplo, las neuronas permanecen luego de la maduración del tejido nervioso en una etapa especial denominada G0, donde las células entrarían como alternativa a G1. En la actualidad es frecuente referirse a este tipo de células como "no cíclicas" o detenidas en G1, ya que no es seguro que las células que no se dividen pasen por un solo estadío.
Etapas y caracterÍsticas
Como ya se mencionó, una célula tipo pasa a lo largo de su vida por etapas (G1, S y G2) antes de dividirse. Las características más relevantes de cada una de las mismas son:
Etapa G1: Esta etapa que sucede a la división celular es la más variable en duración. Las células hijas recientemente originadas presentan una gran actividad metabólica produciéndose un aumento acelerado del tamaño celular. Los organoides de la célula precursora han sido repartidos de manera más o menos equitativa entre las células hijas, deben entonces aumentar de tamaño y también en número para mantener las características de su tipo celular. Se sintetizan así ribosomas y microtúbulos a partir de las proteínas y otras moléculas que la conforman. Los organoides del sistema de endomembranas, aumentan considerablemente de tamaño, ya que ambas células hijas han recibido parte de estos organoides. Sin embargo, pueden ser sintetizados de nuevo en caso de no existir precursores. Esto no ocurre con mitocondrias y cloroplastos que se originan por división de estas estructuras preexistentes. Como se recordará ambos organoides contienen ADN y ribosomas que les permite dividirse de forma relativamente independiente del núcleo celular.
Todos los procesos de síntesis de nuevos organoides o aumento de tamaño de los existentes, son regulados mediante activación de complejos enzimáticos en un momento determinado.
En este período se observa, a su vez, una gran síntesis de ARNm como así también ARNt y ARNr. Estos ácidos serán utilizados para la síntesis de proteínas estructurales, para la construcción y o aumento de los organoides, como así también la producción de enzimas necesarias para dicha síntesis. Cabe destacar que durante este período también se sintetizan las enzimas que serán utilizadas en la etapa siguiente, es decir en la duplicación del ADN, como así también moléculas precursoras de los ácidos nucleicos.
Cuando las células dejan de crecer (si se agotan los nutrientes o por inhibición por contacto) lo hacen en G1. Esto implica que también se sintetizan las sustancias que estimulan o inhiben distintas fases del ciclo celular.
Etapa S: el período S o de síntesis de ADN tiene como característica fundamental la síntesis de nuevo material genético, para que las células hijas tengan la misma dotación. Sin embargo persisten los altos índices de síntesis de ARN para obtener enzimas requeridas en la síntesis de histonas que formarán parte de la macroestructura del ADN y tubulinas relacionadas con el proceso de división celular.
Etapa G2: En esta fase, ya con el ADN duplicado, la célula ensambla las estructuras necesarias para la separación de las células hijas durante la división celular y la citocinesis (separación del citoplasma).
Etapa M: Durante M, la envoltura nuclear se desintegra, la cromatina se condensa en forma creciente hasta ser visible los cromosomas al microscopio óptico. Estos cromosomas formados cada uno por dos cromátidas (cromosomas duplicados) pasaran por cada una de las fases de la división celular (mitosis o meiosis) para concluir con la formación de las células hijas, cada una con una única copia de su ADN (cromosomas sin replicar) , que marcan el inicio de un nuevo ciclo.
Sistema de control del ciclo celular
El sistema de control del ciclo celular es un dispositivo bioquímico compuesto por un conjunto de proteínas reguladoras interactivas: las ciclinas y las quinasas dependientes de ciclinas que inducen y coordinan los procesos básicos del ciclo, como la duplicación de ADN y la división celular, a los que denominamos procesos subordinados.
Durante un ciclo típico, el sistema de control está regulado por factores de retraso que pueden frenar el ciclo en puntos determinados denominados puntos de control. En estos puntos, las señales de retroalimentación que contienen información sobre los procesos subordinados pueden detener momentáneamente el avance del ciclo, evitando el inicio del proceso siguiente antes que el precedente haya terminado. Sobre dichos factores también actúan señales del entorno como puede ser una hormona o un factor de crecimiento.
Una analogía que puede ayudarnos a comprender este mecanismo es comparar al sistema de control del ciclo celular con el funcionamiento de una lavadora automática (1. Alberts y col -pág929-930), el programador de la lavadora sólo avanza a través de los diferentes pasos del ciclo de lavado (etapas del ciclo celular), si recibe determinadas señales. Adentro de la lavadora hay sensores que miden el nivel de agua o jabón que ingresan. Estos sensores envían señales que pueden provocar el retraso o la interrupción del ciclo de lavado. De igual manera en la célula, las señales generadas en los procesos subordinados (por ej. la síntesis de ADN) o por el entorno, detienen el ciclo.
A continuación pasaremos a describir las proteínas reguladoras, el mecanismo de regulación y los puntos de control del ciclo celular.
ProteÍnas reguladoras del Ciclo Celular
El pasaje de una célula a través del ciclo es controlado por proteínas citoplasmáticas. Los principales reguladores del ciclo en células animales son:
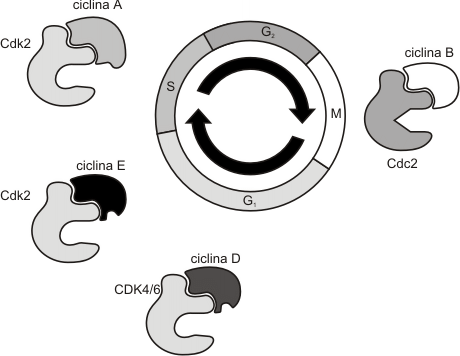
1. Las ciclinas, proteínas que controlan la actividad de sus proteinquinasas dependientes. La concentración de ciclinas varía en forma cíclica, aumentando o disminuyendo durante el transcurso del ciclo celular. Esto se debe a variaciones en la velocidad de degradación de la ciclina, dado que la velocidad de síntesis es casi constante durante todo el ciclo. En los mamíferos existen 6 ciclinas como mínimo, denominadas A, B, C, D, E y F (Fig. 12.4b), pero nosotros las clasificaremos como ciclinas de G1 y ciclinas mitóticas. Las ciclinas G1 se unen a sus quinasas dependientes de ciclinas (Cdk2) durante G1 siendo necesarias para superar el punto de control G1 y pasar a la fase S. Las ciclinas mitóticas se fijan a la quinasa Cdk1 durante G2, siendo necesaria su presencia para que el ciclo supere el punto de control G2 y se inicie la mitosis. (Fig. 12.3 )
2. Las quinasas dependientes de ciclinas (CDK), enzimas que mediante la fosforilación de determinadas proteínas desencadenan los procesos subordinados del ciclo celular. En los mamíferos se conocen 5 CDK las cuales forman tres grupos principales:
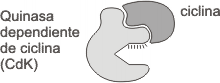
Fig. 12.3 - Complejo ciclina-quinasa dependiente de ciclina activo (ciclina-CDK)
· CDK de G1 (Cdk2)
· CDK de fase S (Cdk2)
· CDK de fase M (Cdk1)
A diferencia de la concentración de ciclinas, la concentración de CDK se mantiene durante todo el ciclo celular, por permanecer constantes tanto la velocidad de síntesis como la de degradación (Fig. 12.4 y 12.5)
Las CDK se activan sólo cuando se unen a las ciclinas para formar complejos, por lo que requieren un nivel umbral para desencadenar la transición a la fase siguiente del ciclo celular.
3. El Complejo Promotor de la Anafase (APC) y otras enzimas proteolíticas. El APC desencadena los eventos que conducen a la destrucción de las cohesinas [1] permitiendo a las cromátidas hermanas separarse e iniciando la degradación de las ciclinas mitóticas.
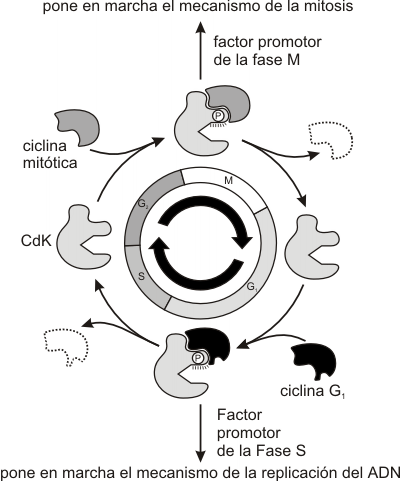
Fig. 12.4 -Generalización del sistema de control del ciclo celular en eucariotas
Mecanismo de regulaciÓn del ciclo celular
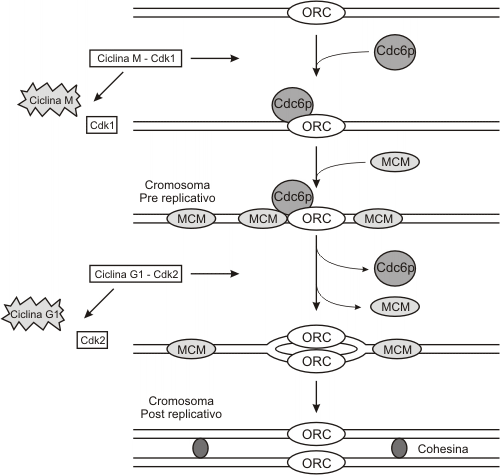
Al finalizar la mitosis aumenta la expresión de la ciclina G1 (E), esta ciclina se unirá a la su quinasa (Cdk2) formando un complejo activo conocido como factor promotor de Fase S (FPS ). Este FPS sólo puede actuar sobre cromosomas en estado Pre-Replicativo. Así se denominan por poseer sobre cada origen de replicación un complejo multiproteico llamado Pre-Replicativo.
Los orígenes de replicación (ORI) se presentan en número de 20 a 80 sobre cada lazo de cromatina y se caracterizan por poseer una secuencia común denominada secuencia de replicación autónoma (ARS) formada por dos secuencias "GAGGC" sobre las que se halla unido a lo largo de todo el ciclo celular, el complejo de reconocimiento del origen de replicación (ORC), uno de los complejos proteícos que forma parte del complejo Pre-Replicativo (PreR). El segundo componente del complejo PreR es la proteína Cdc6p (cell division cycle protein), que se sintetiza en G1 e inserta sobre los orígenes de replicación al último componente, las proteínas de mantenimiento de los minimicrosomas (MCM). (Fig. 12.6)
El nivel creciente de FPS al inicio de la fase S induce la apertura de los orígenes de replicación, activando a las moléculas responsables de la síntesis de ADN e induciendo la separación del complejo Pre-R del componente Cdc6p y MCM. Separados estos componentes, se inicia la síntesis, y por lo tanto el FPS no se requiere más, siendo su componente lábil, la ciclina de G1, degradada en los proteosomas.
Los cromosomas a partir de este momento se denominarán cromosomas Post-Replicativos (sólo presentan asociado a los orígenes de replicación el ORC). Los cromosomas se mantendrán en estado Post-R hasta el inicio de la anafase.
Degradadas las ciclinas G1, el nivel de ciclinas mitóticas aumenta.
Un nuevo participante entra al ciclo, el complejo promotor de la mitosis, FPM, formado por las ciclinas mitóticas más las quinasas dependientes de ciclinas de M (Cdk1). Éste inicia el ensamblado del huso mitótico, la desintegración de la envoltura nuclear y la condensación de los cromosomas, al inducir la fosforilación de diferentes sustratos como las láminas nucleares, conduciendo a la célula a la metafase.
A esta altura del ciclo, el FPM activa el complejo promotor de la Anafase, APC, que permite la separación de las cromátides hermanas y su migración a los polos (anafase). Así se completa la mitosis, se destruyen las ciclinas de fase M y se activan las ciclinas de G1 para el próximo ciclo celular.
Control de calidad del Ciclo Celular (Fig. 12.7)
Durante el ciclo celular, la célula pasa al menos tres puntos de control (checkpoints):
· Punto de control G1, en este punto el sistema de control de la célula pondrá en marcha el proceso que inicia la fase S. El sistema evaluará la integridad del ADN (que no este dañado), la presencia de nutrientes en el entorno y el tamaño celular. Aquí es donde generalmente actúan las señales que detienen el ciclo (arresto celular) .
· Punto de control G2, en él se pone en marcha el proceso que inicia la fase M. En este punto, el sistema de control verificará que la duplicación del ADN se halla completado (que no este dañado), si es favorable el entorno y si la célula es lo suficientemente grande para dividirse.
· Punto de control de la Metafase o del Huso, verifica si los cromosomas están alineados apropiadamente en el plano metafásico antes de entrar en anafase. Este punto protege contra pérdidas o ganancias de cromosomas, siendo controlado por la activación del APC.
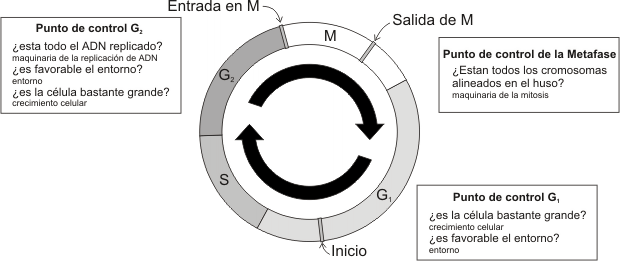
Fig. 12.7 - Puntos de Control e Ingreso de la información Reguladora al Sistema de Control del Ciclo Celular
Proteína p53, el guardián del genoma
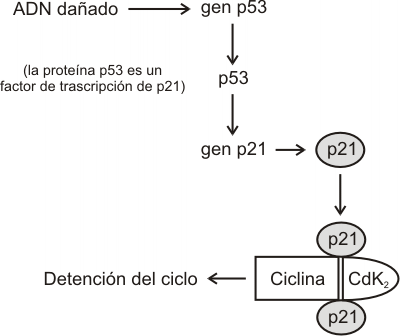
Como hemos mencionado en los párrafos precedentes, tanto en el punto de control G1 como G2 se verifica la integridad del ADN. Ante la presencia de ADN dañado se genera una señal que retrasa la entrada en fase M. El mecanismo depende de una proteína llamada p53, que se acumula en la célula en respuesta a las alteraciones de ADN, deteniendo el sistema de control en G1 y por lo tanto impidiendo la posterior entrada en mitosis. El gen p53 es uno de los genes supresores de tumores más conocidos, que no sólo detiene el ciclo (arresto celular), sino también participa en la apoptosis (muerte celular programada) forzando a las células al suicidio cuando el daño en el ADN es irreparable.
Las células que presentan los dos alelos del gen p53 mutados, tendrán proteína p53 no activa y por lo tanto continuarán dividiéndose a pesar del daño en su genoma, por lo tanto desarrollarán cáncer. Las mutaciones del gen p53 presenta una alta incidencia en la mayoría de los cánceres humanos.
¿Cómo actúa la p53?
Cuando el ADN presenta un daño "limitado", aumentan los niveles de proteína p53. Dicha proteína activa la transcripción del gen p21, que codifica a la proteína p21. Esta última proteína ejerce su efecto inhibidor uniéndose al complejo ciclina-Cdk2 y deteniendo el ciclo. Cuando el ADN es reparado, la proteína p53 se libera del promotor del gen p21, provocando el descenso en los niveles de p21. Esto permite restaurar la actividad del complejo ciclina-Cdk2.
Oncogenes y CÁncer
Los genes supresores de tumores, codifican para productos celulares que inhiben la proliferación celular. Para impedir el efecto protector que ejercen sobre el genoma, se requiere la mutación de sus dos alelos.
Los genes conocidos como protooncogenes codifican proteínas que estimulan la división celular, por ejemplo, factores del crecimiento o receptores de factores del crecimiento.
La mutación de uno de los dos alelos que codifican para un protooncogen, lo transforma en un oncogen capaz de originar productos celulares que estimulan la división celular de forma incontrolada conduciendo al cáncer, con alteración de los mecanismos de control del ciclo celular.
En la siguiente tabla se mencionan a titulo informativo los oncogenes y genes supresores de tumores mejor conocidos por su expresión durante el ciclo celular.
|
Tabla 12.1 - Algunos genes relacionados con cÁnceres en humanos |
||
|
Oncogenes |
Genes para factores de crecimiento o sus receptores |
|
|
PDGF |
Codifica el Factor de crecimiento derivado de las plaquetas. Responsable de glioma (un cáncer del cerebro) |
|
|
erb-B |
Codifica al Factor de crecimiento epidérmico. Relacionado con gliobastoma (cáncer del cerebro) y cáncer de mama. |
|
|
erb-B2 |
Codifica receptor de factor de crecimiento. Relacionado con cáncer de mama, glándulas salivales y ovario. |
|
|
RET |
Codifica receptor de Factor del crecimiento. Relacionado con cáncer de tiroides. |
|
|
Oncogenes |
Genes para transductores citoplasmáticos en vías estimuladoras |
|
|
Ki-ras |
Responsable de cáncer de pulmón, ovario, colon y páncreas. |
|
|
N-ras |
Relacionado con leucemias. |
|
|
Genes para factores de transcripción que activan genes promotores del crecimiento |
||
|
c-myc |
Relacionado con leucemias y cánceres de estómago, pulmón y mamas |
|
|
N-myc |
Relacionado con neuroblastoma (cáncer de células nerviosas) y glioblastoma |
|
|
L-myc |
Relacionado con cáncer de pulmón. |
|
|
Genes para otros tipos de moléculas |
||
|
Bcl-2 |
Codifica para una proteína que normalmente bloquea la apoptosis. Relacionado con linfoma de células foliculares B. |
|
|
Bcl-1 |
También llamado PRADI. Codifica la ciclina D1, un ciclina reguladora del ciclo celular. Relacionada con cáncer de mama, cabeza y cuello. |
|
|
MDM2 |
Codifica un antagonista de la proteína p53. Participa en sarcomas y otros cánceres. |
|
|
Genes Supresores de Tumores |
Genes de proteínas citoplasmáticas |
|
|
APC |
Relacionada con cáncer de colón y estómago. |
|
|
DPC4 |
Codifica para una molécula transductora en una vía que inhibe la división celular. Relacionada con cáncer pancreático. |
|
|
NF-1 |
Codifica para una proteína que inhibe a la proteína Ras. Relacionada con neurofibroma y feocromocitoma (cáncer del sistema nervioso periférico) y leucemia mieloide. |
|
|
NF-2 |
Relacionado con meningioma y ependinoma (encéfalo) y schwanoma (nervios periféricos) |
|
|
Genes de proteínas nucleares |
||
|
MTS1 |
Codifica para la proteína p16, uno de los frenos del sistema de control del ciclo celular. Relacionada con muchos cánceres. |
|
|
RB |
Codifica para la proteína RB (retinoblastoma). Esta proteína es uno de los principales frenos en el ciclo celular. |
|
|
p53 |
Codifica para la proteína p53, la cual detiene la división celular e induce a las células anormales al suicidio (apoptosis). Relacionado con la mayoría de los cánceres . |
|
|
WT1 |
Relacionado con el Tumor de Wilms del riñón. |
|
|
Genes que codifican proteínas de ubicación aún no determinada |
||
|
BRCA1 |
Relacionado en cánceres de mama y ovario. |
|
|
BRCA2 |
Relacionado con cáncer de mama. |
|
|
VHL |
Relacionado con cáncer de células renales. |
|
CaracterÍsticas de la duplicaciÓn del ADN
Aunque los principios generales de la duplicación o replicación del ADN son sencillos y pueden considerarse como consecuencia directa de su estructura, el proceso requiere una maquinaria compleja que contiene una gran cantidad de enzimas y proteínas que actúan en conjunto.
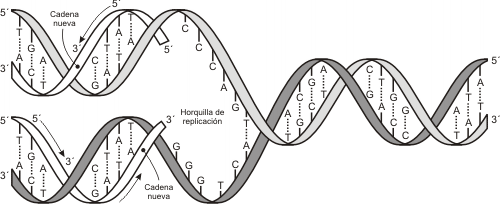
Fig. 12.9 - La duplicación del ADN se produce previo desenrollamiento de las dos cadenas de la doble hélice, usando cada una como molde para sintetizar las nuevas cadenas.
1. MÚltiples puntos de origen
Los cromosomas eucariontes tienen una gran cantidad de ADN, el cual se halla contenido en dos moléculas lineales, una para cada cromátide. Si estas moléculas se replicasen a partir de un sitio único de origen, la etapa “S” de la Interfase sería extremadamente larga. Las células eucariontes resuelven este problema disponiendo de múltiples sitios de origen de la replicación en cada cromosoma. En ellos, el ADN presenta secuencias especiales de nucleótidos. Además todos los orígenes tienen en común secuencias conservadas de aproximadamente doce pares de nucleótidos, llamados ARS (autonomus replication secuence).
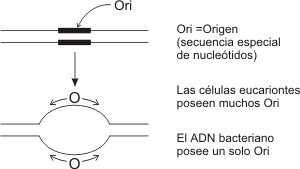
Fig. 12.10 - La replicación siempre comienza en los sitios de origen en cada cromosoma. En ellos el ADN presenta secuencias especiales de nucleótidos
2. Desenrollamiento de las cadenas de ADN
En cada cromosoma, las dos cadenas del ADN se encuentran arrolladas una a la otra como los hilos de una soga. Si tratamos de separarlas, la soga debe apretarse más en las vueltas restantes o girar. Algo similar ocurre en el ADN, cuando las cadenas complementarias se separan para iniciar la duplicación. En ese momento aumenta la tensión torsional en el sector no duplicado de la doble hélice.
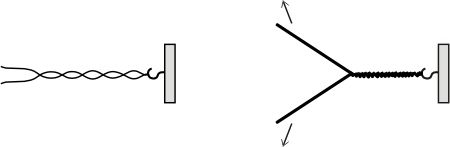
Fig. 12. 11 - Separación progresiva de las dos cadenas de ADN a nivel de la horquilla de replicación y su posible consecuencia biológica.
El desenrollamiento es realizado por las enzimas ADN-helicasas, las cuales recorren la hélice desenrollando las hebras del ADN a medida que avanzan. Una vez separadas, las cadenas se combinan con las proteínas SSB, llamadas también proteínas desestabilizadoras, que evitan el autoapareamiento entre las bases complementarias libremente expuestas.
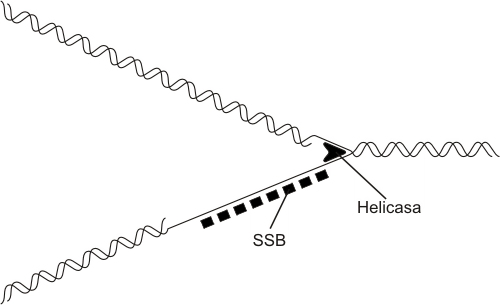
Fig. 12.12 - Cadenas adelantada y retrasada del ADN durante la replicación. La helicasa separa a las dos cadenas del ADN y las proteínas SSB evitan autoapareamientos entre las bases complementarias libremente expuestas en la cadena atrasada.
A medida que la enzima helicasa abre la doble hélice, dos enzimas complementarias: la topoisomerasa I y la topoisomerasa II o girasa, van disminuyendo la tensión torsional acumulada por el superenrollamiento en el sector no replicado de la doble hélice.
La topoisomerasa I primero corta una de las cadenas del ADN, luego la cadena cortada gira una vuelta en torno a su propio eje y finalmente vuelve a unir los extremos cortados.
La topoisomerasa II corta las dos cadenas, las cuales luego de girar una vuelta alrededor del eje de la doble hélice, restablecen sus uniones.
Ambas enzimas utilizan energía del ATP y se comportan como nucleasas (cortando las cadenas de ADN) y luego como ligasas (restableciendo las uniones fosfodiéster).
Las topoisomerasas I y II se diferencian no sólo porque la primera corta una de las cadenas y la segunda corta las dos, sino también porque la topoisomerasa I realiza desenrollamientos de corto alcance y la girasa abarca una extensión de ADN bastante mayor.
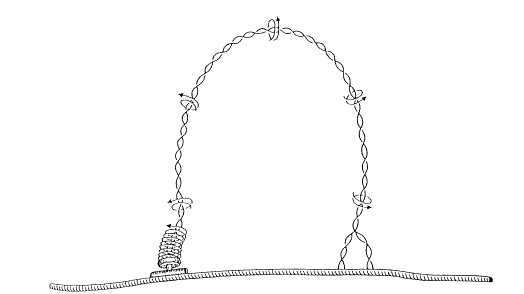
Fig. 12.12 - Efecto hipotético de la topoisomerasa II para evitar el superenrollamiento que se produce en el ADN como consecuencia de la separación progresiva de sus cadenas.
3. La duplicaciÓn del ADN es bidireccional
Al abrirse la doble hélice se forma una “burbuja” de replicación, cuyo tamaño aumenta a medida que avanza la separación de las dos cadenas del ADN, fenómeno que se produce en ambos extremos de la burbuja en forma simultánea. Se establece de este modo, en cada uno de los extremos, una estructura en forma de Y, a la que llamamos horquilla de replicación, cuyos brazos representan a las cadenas ya separadas de ADN y el tronco la doble hélice en vías de separación. Así cada burbuja tiene dos horquillas de replicación que a partir de un punto de origen común avanzan en direcciones opuestas. Las horquillas desaparecen cuando se van integrando a las burbujas contiguas. Las horquillas que recorren los telómeros desaparecen cuando se separa el último par de nucleótidos.
El segmento de ADN que se sintetiza a partir de un origen de replicación (con sus dos horquillas), lo llamamos replicón. De este modo la replicación del ADN termina cuando se ensamblan los sucesivos replicones. Esto permite que el ADN se sintetice en un tiempo bastante breve para el ciclo de vida de una célula.
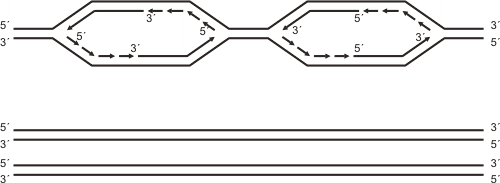
Fig. 12. 14 - Esquema que muestra dos replicones contiguos y los puntos donde se origina la replicación (flechas). Puede observarse el carácter bidireccional de la replicación y los sectores donde el ADN se sintetiza en forma continua y discontinua.
4. La sÍntesis de ADN es discontinua
Una de las características
de las ADN-polimerasas es que sólo pueden actuar en dirección 5’![]() 3’, por agregado de nucleótidos en el extremo 3’ de
las cadenas nuevas. Como vimos, a medida que se separan las cadenas progenitoras
en la horquilla de replicación, una presenta sus nucleótidos en dirección
5’
3’, por agregado de nucleótidos en el extremo 3’ de
las cadenas nuevas. Como vimos, a medida que se separan las cadenas progenitoras
en la horquilla de replicación, una presenta sus nucleótidos en dirección
5’![]() 3’ y la otra en
dirección 3’
3’ y la otra en
dirección 3’![]() 5’. De manera que la primera
al ser copiada debería formar una cadena hija en sentido 3’
5’. De manera que la primera
al ser copiada debería formar una cadena hija en sentido 3’![]() 5’, algo que las polimerasas no pueden hacer.
5’, algo que las polimerasas no pueden hacer.
Las células solucionan
esta situación utilizando estrategias distintas en la construcción de
cada una de las nuevas cadenas. La cadena hija que se formó en dirección
5'![]() 3’ se construye
en forma continua mediante el agregado de nucleótidos en el extremo 3’
a medida que avanza la horquilla de replicación. En cambio la otra cadena
hija es sintetizada de manera discontinua, en pequeños tramos, llamados
fragmentos de Okazaki, los cuales se unen entre sí a medida que se sintetizan,
por acción de la enzima ADN-ligasa.
3’ se construye
en forma continua mediante el agregado de nucleótidos en el extremo 3’
a medida que avanza la horquilla de replicación. En cambio la otra cadena
hija es sintetizada de manera discontinua, en pequeños tramos, llamados
fragmentos de Okazaki, los cuales se unen entre sí a medida que se sintetizan,
por acción de la enzima ADN-ligasa.
Por lo expuesto, podemos decir que la síntesis del ADN es un proceso bidireccional, no sólo porque se produce en dos direcciones divergentes a partir de una misma burbuja de replicación, sino también porque las cadenas de la doble hélice son sintetizadas en direcciones opuestas.
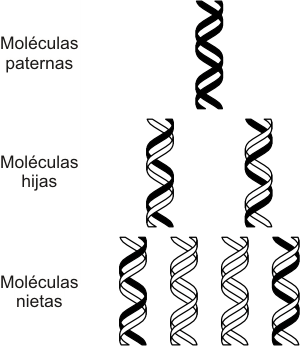
Fig. 12. 15 - Replicación semiconservativa del ADN.
5. Modelo semiconservativo
Como las nuevas hélices de ADN están formadas por una cadena original (preexistente) que sirvió de molde y una cadena nueva (recién sintetizada) decimos que el mecanismo de replicación es semiconservativo.
Inicio de la síntesis de ADN
Para iniciar la síntesis de las cadenas complementarias se requiere además del ADN molde un cebador o primer, que consiste en una pequeña cadena de ARN de unos diez nucleótidos de largo. La síntesis del cebador es catalizada por una enzima llamada ARN-primasa y el cebador queda unido al ADN temporariamente. Una vez sintetizado el cebador la síntesis continúa si la ADN-polimerasa tiene disponibles suficientes desoxirribonucleótidos trifosfatados (dATP, dGTP, dCCP y dTTP). Éstos se agregan en la cadena nueva de acuerdo a la secuencia de nucleótidos de la cadena que sirve de molde.
Gran parte de la energía requerida durante la replicación la aportan los desoxirribonucleótidos trifosfatados, que hidrolizan los últimos dos grupos fosfato (liberando energía) cuando se unen entre sí.
Iniciada la síntesis de ADN a partir del cebador, la ADN-polimerasa a (alfa) es reemplazado por la ADN-polimerasa e (epsilon), enzima de alta procesividad (más rápida), que elonga la hebra continua, mientras que la ADN-polimerasa d (delta) se encarga de elongar los fragmentos de Okazaki de la hebra discontínua.
Las ADN-polimerasa e (epsilon) y d (delta) catalizan la síntesis de las cadenas nuevas agregando nucleótidos en el extremo 3’ de las hebras en formación. Mientras tanto los cebadores de la cadena rezagada son removidos por la actividad nucleasa reparadora de la ADN-polimerasa d (delta) y su lugar es reemplazado por un fragmento de ADN equivalente. Como vimos la cadena continua requiere un solo cebador que se forma a comienzo de la replicación, en cambio la cadena discontinua necesita muchos cebadores, uno para cada fragmento de Okazaki. Cabe señalar que las ADN-polimerasas d (delta) y e (epsilon) son sostenidas por un anillo proteico llamado PCNA (Proliferating Cell Nuclear Antigen) mientras realiza el deslizamiento por la cadena de ADN. Está asociación entre polimerasas y PCNA hace posible el aumento en la velocidad de síntesis. Al inicio, la síntesis es lenta debido a que el complejo ADN polimerasa a (alfa)-primasa no se asocia al anillo de PCNA.
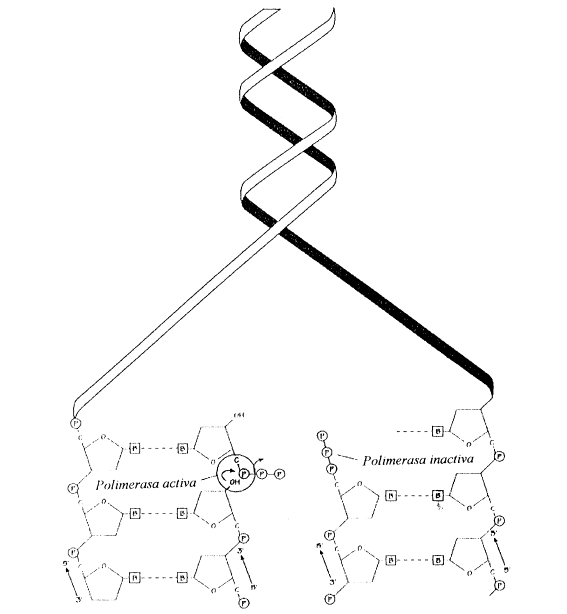
Fig. 12.16 - Doble hélice de ADN desenrrollándose. Cada cadena servirá de molde para la síntesis de cadenas nuevas
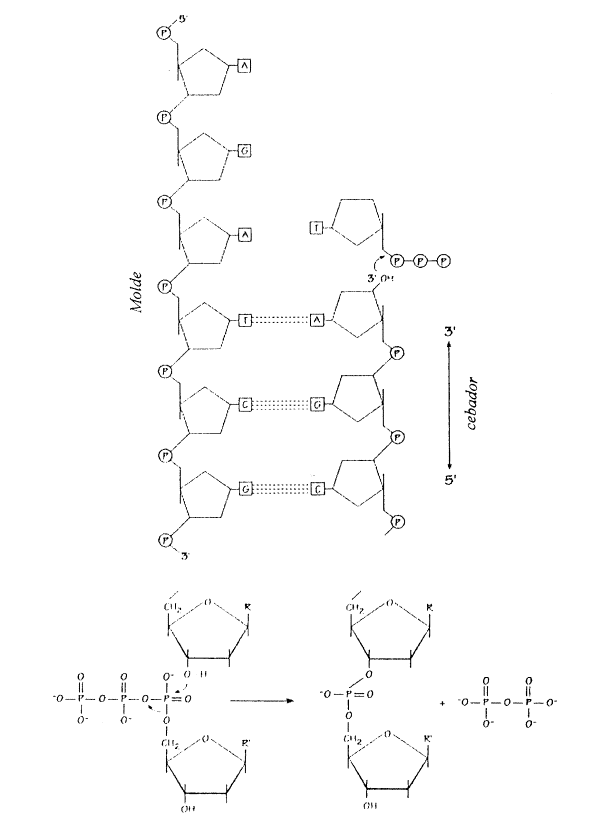
Fig. 12.17 - La energía para el proceso de replicación la proveen los mismos desoxirribonucleótidos trifosfatados, con la hidrólisis de los últimos dos grupos fosfato-
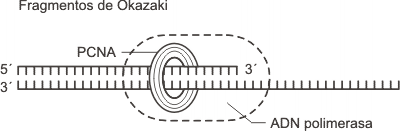
Fig. 12. 18 - Unión del aro de PCNA a la ADN polimerasa
Los fragmentos de Okazaki alcanzan una longitud de 200 nucleótidos. Una vez completa la síntesis de un fragmento ambas enzimas se liberan y vuelven juntas hacia el ángulo de la horquilla de replicación, dejando atrás los 200 nucleótidos del segmento que acaban de sintetizar y otros tantos del ADN molde que se usará para copiar el próximo fragmento de Okazaki.
Como ocurre con la polimerasa d en movimiento, la ADN-polimerasa a no se desprende del ADN debido a que se le asocia un aro de PCNA. Luego el cebador es hidrolizado por una nucleasa, la que se encarga también de reparar errores (segundo sistema de reparación) y los huecos son rellenados con desoxirribonucleótidos por la ADN-polimerasa b (beta). Finalmente los fragmentos de Okazaki son unidos por la ADN-ligasa.
La discontinuidad de la síntesis hace que la hebra mal orientada crezca más lentamente que la otra, que lo hace en forma continua, por esta razón se les asignó el nombre de cadena rezagada y cadena adelantada respectivamente.
Replicación de la heterocromatina
La heterocromatina por estar muy compactada se replica tardíamente durante la fase “S” del ciclo celular.
Síntesis de histonas
Hemos señalado que el ADN se replica en forma semiconservativa, es decir que las cadenas de la doble hélice progenitora, al separarse para su replicación, se comparten por igual en ambos cromosomas hijos. En cambio no se sabe como se segregan las histonas. Es posible que al cabo de la replicación sean heredadas totalmente por uno de los cromosomas hijos, en este caso el otro cromosoma hijo debe procurarse de la totalidad de histonas nuevas.
En su mayor parte las histonas se sintetizan durante la fase “S” de la interfase y se incorporan al cromosoma apenas es duplicado el ADN
Replicación en Procariontes
Muchas de las características esenciales de la duplicación del ADN son universales, aunque existen algunas diferencias entre procariontes y eucariontes debido a que su material genético está organizado de manera distinta.
En las bacterias casi todo el ADN se encuentra formando una cadena circular, en cambio en los eucariontes cada cromosoma no duplicado contiene una cadena lineal asociada a una gran cantidad de proteínas y algo de ARN.
En procariontes al existir un único punto de origen se forma sólo un ojal de replicación. Aquí actúan tres ADN-polimerasas:
ADN-polimerasa I, es la que elimina el cebador y reemplaza los ribonucleótidos de éste por desoxirribonucleótidos, rellenando los huecos dejados por la ADN-polimerasa II. Adiciona 10 nucleótidos/seg.
ADN-polimerasa II, tiene actividad de nucleasa, es decir que retira nucleótidos de la cadena de ADN en los sitios donde se produjeron errores o desemparejamientos.
ADN-polimerasa III, es la enzima responsable de alargar las cadenas en el proceso de replicación. Adiciona 500 nucleótidos/seg.
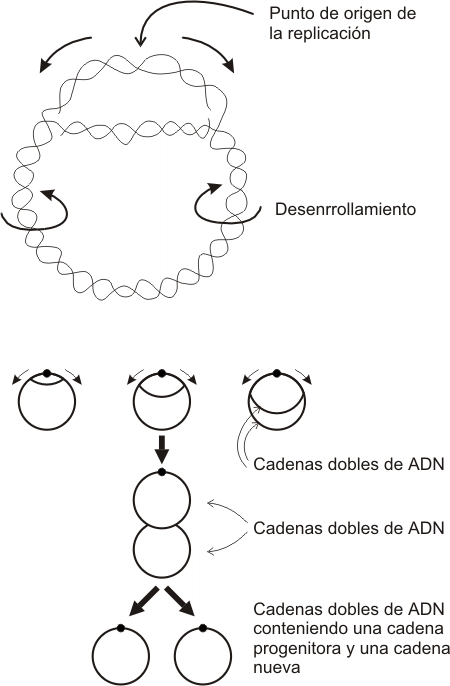
Fig. 12.19 - Mecanismo de replicación del ADN en células procariontes
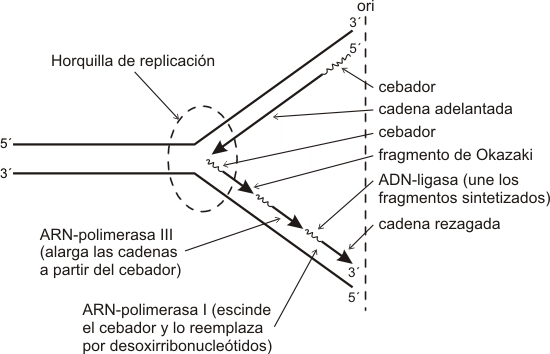
Fig. 12.20 - Mecanismo de replicación en procariotas
|
Cuadro
12.1 - MÚltiples funciones de las ADN-polimerasa en E. coli
|
|
|
Poli. I y Poli. III |
·
Síntesis de ADN en sentido 5’ ·
Exonucleasa en sentido 3’ ·
Corrección de errores, removiendo nucleótidos en
sentido 5’ |
|
Poli. I |
·
Exonucleasa en sentido 5’ |
|
Cuadro 12.2 - Principales enzimas que actuan en la replicaciÓn |
|
|
Enzimas |
Reacciones que catalizan |
|
ADN-polimerasa I (procariontes) |
Elimina el cebador, reemplazando los ribonucleótidos por desoxirribonucleótidos. Rellena los huecos del ADN. |
|
ADN-polimerasa II (procariontes) |
Nucleasa, corrige errores. |
|
ADN-polimerasa III (procariontes) |
Principal enzima de la replicación. Sintetiza la cadena nueva a partir del cebador. Realiza corrección de pruebas. |
|
Complejo ADN-polimerasa a-primasa (eucariontes) |
La primasa del complejo sintetiza un "primer" de ARN y a partir de dicho primer, la polimerasa a del complejo inicia la síntesis de ADN. |
ADN-polimerasa e (eucariontes) |
Polimerización de desoxirribonucleótidos de la cadena adelantada. |
ADN-polimerasa d (eucariontes) |
Polimerización de desoxirribonucleótidos de la cadena rezagada. |
ADN-polimerasa b, q, l |
Reparación del ADN. |
|
ADN-polimerasa g (eucariontes) |
Duplicación y reparación del ADN mitocondrial. |
|
Helicasas |
Rompen los puentes de hidrógeno, separando las cadenas del ADN. |
|
Primasas |
Sintetizan el primer o cebador. |
|
Proteínas SSB |
Se extienden sobre las hebras del ADN evitando autoapareamientos entre las bases libremente expuestas |
|
Topoisomerasas I y II |
Corrigen el superenrollamiento |