NATURALEZA MOLECULAR DEL GEN Y DEL GENOMA
INTRODUCCIÓN
Desde que el hombre se ha dedicado al cultivo o ha criado animales, resulta obvio que cada semilla o cada óvulo fecundado ha de contener un plan o diseño para el desarrollo del organismo. En la época moderna, la ciencia de la genética se desarrolló alrededor de la premisa de que existen unos elementos invisibles portadores de información, denominados genes, que son distribuidos a las células hijas cuando la célula madre se divide. Por consiguiente, antes de dividirse una célula debe de hacer una copia de sus genes para poder ceder a sus células hijas una colección completa de ellos. Los genes de los espermatozoides y de los óvulos transmiten la información genética de una generación a la siguiente.
Hacia finales del siglo XIX, los biólogos habían reconocido ya que los transportadores de la información hereditaria eran los cromosomas que resultan visibles en el núcleo cuando una célula comienza a dividirse. Pero la evidencia de que es el ADN de estos cromosomas la sustancia de la que están formados los genes, se obtuvo mucho más tarde a partir de estudios sobre microorganismos. Hasta entonces se había creído que sólo las proteínas presentaban una complejidad conformacional suficiente como para ser portadoras de la información genética. El modelo del ADN propuesto por Watson y Crick en 1953 brindó una explicación satisfactoria de la forma en que pueden operar los procesos relacionados con una molécula depositaria de la información genética: el almacenamiento de información, su replicación, su expresión, la mutación y la recombinación.
El presente capítulo tratará acerca de la naturaleza de los genes y su expresión en dos pasos: la transcripción y la traducción; asimismo nos ocuparemos de los mecanismos de regulación que controlan la expresión genética.
EL CONCEPTO DE GEN
El genoma es el conjunto de genes de una especie. Pero ¿qué es un gen?. En principio es una unidad informativa discreta, responsable de una característica transmisible, vg. el color de ojos, la textura de la semilla, la longitud del tallo. Este es el concepto mendeliano del gen, acuñado en el siglo XIX, cuando aún se desconocía su verdadera naturaleza química. Esta definición del gen sigue siendo útil en determinado contexto, con la salvedad de que hoy identificamos a dicha unidad de información con un fragmento de ADN localizado en determinado lugar de un cromosoma. Una segunda concepción del gen surgió cuando los genetistas demostraron de forma concluyente que los genes especifican la estructura de las proteínas individuales. A principios de los años 1950 se llegó a conocer la secuencia de aminoácidos de la proteína insulina y se descubrió que cada proteína consiste en una secuencia de aminoácidos típica, de la cual, se supuso, dependerían sus propiedades. Al mismo tiempo se correlacionaron mutaciones (alteraciones en la secuencia de nucleótidos del ADN) con alteraciones de la secuencia de aminoácidos en las proteínas. Resultó evidente que la secuencia del ADN especifica, mediante algún código, la secuencia proteica. Un gen es, entonces, una secuencia de ADN con la información necesaria para la síntesis de una proteína particular.
Dado que el ADN es una molécula relativamente inerte, su información se expresa indirectamente, a través de otras moléculas. El ADN dirige la síntesis de proteínas y éstas determinan las características físicas y químicas de la célula.
Como ya adelantáramos, las instrucciones genéticas contenidas en el ADN se expresan en dos pasos. El primero de ellos, la transcripción, consiste en la síntesis de ARN a partir del ADN. El ARN contiene toda la información de la secuencia de bases del ADN de la que ha sido copiado. El segundo paso de la expresión genética es la traducción, momento en el cual el ARN ejecuta las instrucciones recibidas cristalizándolas en la síntesis de una proteína específica.
Existen diversos tipos de ARN: el ARNm (mensajero), el ARNr (ribosómico), el ARNt (de transferencia) y los ARN pequeños. De todos ellos, tan sólo el ARNm es portador de información acerca de la secuencia aminoacídica de una proteína; sin embargo todos ellos son transcriptos de ADN. Por lo tanto, resulta necesario revisar la segunda definición del gen.
Hoy se acepta que un gen es una secuencia de ADN transcripta que genera un producto con función celular específica.
Cabe aclarar, no obstante, que esta definición no es completamente satisfactoria, en la medida en que existen regiones reguladoras de los genes, que no se transcriben, y otras regiones (llamadas intrones) que se transcriben pero se eliminan sin cumplir ninguna función aparente.
LA ORGANIZACIÓN DEL GENOMA
Con la excepción de ciertos virus, que contienen un genoma de ARN, el resto de los genomas utiliza el ADN como depositario de la información genética.
Sin embargo, debemos señalar algunas diferencias significativas en cuanto a la organización del genoma en procariontes y eucariontes.
· En primer lugar, el ADN procarionte se presenta como una única molécula circular, en tanto el ADN eucarionte es de estructura lineal. Además las células eucariotas poseen usualmente más de una molécula de ADN en sus núcleos. Cada molécula corresponde a un cromosoma, cuyo número es constante para todas las células de una misma especie (con excepción de las gametas).
· El ADN eucarionte, por otro lado, se halla asociado íntimamente a diferentes proteínas, entre las cuales las histonas juegan el papel más importante en lo que respecta al empaquetamiento del ADN. Esta asociación a histonas no se verifica en el ADN procarionte, por lo cual se lo ha denominado “ADN desnudo”.
En las células eucariotas los cromosomas están confinados en el compartimiento nuclear, donde tiene lugar la transcripción, mientras que la traducción se localiza en el citoplasma; por lo tanto, ambos procesos se encuentran separados espacial y temporalmente. Esto permite que los transcriptos de ARN experimenten en el núcleo un proceso de maduración previo a la traducción. En las células procariotas, donde no existe la envoltura nuclear, el ADN está en contacto directo con el citosol, y los procesos de transcripción y traducción no se hallan separados en espacio ni en tiempo. Los ARNs no son sometidos a modificaciones postranscripcionales.
· Los eucariontes tienen genomas mucho más grandes que los procariontes. Aunque el valor C (cantidad haploide de ADN de una especie) es muy variable entre los mismos eucariontes (ver tabla 1), es siempre notablemente mayor que el de los procariontes. No necesariamente un mayor valor C refleja una mayor complejidad genética (véase en tabla 11.1 valores de Salamandra y Homo sapiens).
· En los procariontes se da una máxima economía de información: en casi todos los casos cada cromosoma contiene una sola copia de cualquier gen particular, y, con excepción de las secuencias reguladoras y señaladoras, prácticamente se expresa todo el ADN. En cambio, en toda célula eucariota parecería haber un gran exceso de ADN, o por lo menos de ADN cuyas funciones se desconocen por completo. Por ejemplo, la estimación del ADN “innecesario” en los seres humanos llega a ser tan elevada como el 95% del genoma.

EL CÓDIGO GENÉTICO
Hemos visto que los genes son segmentos de ADN situados en los cromosomas, que se comportan como unidades de transcripción. Hemos señalado también que muchos de los ARN transcriptos son productos celulares finales con función propia (al margen de las modificaciones postranscripcionales que requieran para completar su maduración), es decir que, en alguna medida, la transcripción es un paso terminal de la expresión de ciertos genes. Sin embargo, sabemos que muchos otros genes contienen la información necesaria para especificar la secuencia de aminoácidos de las tantas proteínas que una célula es capaz de sintetizar. En estos casos, la transcripción es tan sólo el primer paso de la expresión genética. Los ARN obtenidos, ARN mensajeros, son, como su nombre lo indica, meros transportadores de información que aún debe ser decodificada, información que debe traducirse en la síntesis de una proteína. La traducción es el segundo paso de la expresión genética.
![]()
¿De qué manera la estructura de una molécula de ARNm lleva implícita la información para construir una proteína? La información reside en la secuencia de bases y está “escrita” en un código propio al que llamamos código genético.
Muchas de las características del código genético fueron anticipadas en forma teórica a partir del descubrimiento del ARNm. Pero en el año 1961, Nirenberg y Matthaei desarrollaron una técnica que abrió el camino para que en los siguientes cuatro años el código genético fuera enteramente descifrado.
Podemos pensar al código genético como un idioma. Los idiomas utilizan una cierta cantidad de letras, éstas se combinan para formar palabras, y cada palabra tiene un significado, designa a un objeto particular. En el código genético están presentes todos estos elementos: las letras son las 4 bases que forman las cadenas de ARN (A, U, C, G); las palabras son siempre agrupaciones de 3 letras o tripletes de bases, llamadas codones en la molécula del ARNm, y los objetos designados por dichas palabras son cada uno de los 20 aminoácidos que componen las proteínas.
¿Por qué las palabras-codones se forman con 3 bases? Si cada palabra constara de 1 base, habría 4 palabras, y si se formara con 2, las palabras posibles serían 16. En ninguno de los casos alcanzarían para designar a todos los aminoácidos. Pero se pueden obtener 64 combinaciones diferentes si las bases se combinan de a 3; 64 codones son más que suficientes para nombrar a los 20 aminoácidos.
¿Cuál es la función de los 44 codones restantes? Así como en cada idioma existen palabras distintas con un mismo significado, el código genético emplea codones diferentes para nombrar a un mismo aminoácido. La mayoría de los aminoácidos están codificados por más de 1 codón: existen codones sinónimos.
La presencia de codones sinónimos en el código genético habla de una degeneración, característica que, lejos de resultar desventajosa, reporta a las células sus beneficios. Los codones sinónimos son muy similares entre sí, por lo tanto la sustitución de una sola base en un codón frecuentemente da por resultado otro codón que especifica al mismo aminoácido. Por otra parte, aminoácidos de propiedades semejantes son codificados por codones semejantes. Si en una proteína se reemplaza un aminoácido hidrófobo por otro de iguales características a consecuencia de una mutación, las chances de que el cambio sea viable son mayores.
Esta flexibilidad del código no debe confundirse con ambigüedad: el código no es ambiguo, en tanto cada codón especifica a uno y sólo a un aminoácido. Así no da lugar a error en el momento de ser traducido.
Los codones que codifican aminoácidos suman en total 61. Los 3 codones que no especifican ningún aminoácido, UGA, UAG y UAA, actúan como señales de terminación en la traducción o síntesis de proteínas. Son llamados codones de terminación o stop.
La tabla del código que se halla a continuación es válida para los seres vivos más diversos: el hombre, las bacterias, las levaduras, las plantas. El código genético es universal, lo cual da prueba de que todos los organismos comparten un mismo origen. Una de las contadas excepciones a la universalidad del código es el ADN mitocondrial, en el cual algunos codones son leídos de manera diferente.
|
SEGUNDA LETRA |
||||||||
|
U |
C |
A |
G |
|||||
|
PRIMERA LETRA |
U |
UUU fenilalanina UUC fenilalanina UUA leucina UUG leucina |
UCU serina UCC serina UCA serina UCG serina |
UAU tirosina UAC tirosina UAA stop UAG stop |
UGU cisteína UGC cisteína UGA stop UGG triptófano |
U C A G |
TERCERA LETRA |
|
|
C |
CUU leucina CUC leucina CUA leucina CUG leucina |
CCU prolina CCC prolina CCA prolina CCG prolina |
CAU histidina CAC histidina CAA glutamina CAG glutamina |
CGU arginina CGC arginina CGA arginina CGG arginina |
U C A G |
|||
|
A |
AUU isoleucina AUC isoleucina AUA isoleucina AUG metionina |
ACU treonina ACC treonina ACA treonina ACG treonina |
AAU asparagina AAC asparagina AAA lisina AAG lisina |
AGU serina AGC serina AGA arginina AGG arginina |
U C A G |
|||
|
G |
GUU valina GUC valina GUA valina GUG valina |
GCU alanina GCC alanina GCA alanina GCG alanina |
GAU aspartato GAC aspartato GAA glutamato GAG glutamato |
GGU glicina GGC glicina GGA glicina GGG glicina |
U C A G |
|||
El marco de lectura
¿De qué manera se leen los codones durante la síntesis proteica?
Consideremos la siguiente secuencia de bases de un ARNm:
5’-----GUUCCCAUA----3’
Si comenzamos la lectura en la G situada hacia el extremo 5’, este mensaje podría ser traducido como una secuencia de tres aminoácidos (las palabras del código se leen de corrido, sin ningún “signo de puntuación” entre ellas):
Valina - prolina - isoleucina
¿Cuál sería la traducción del mensaje si en cambio la lectura se iniciara en la primera U desde el extremo 5’?
fenilalanina - prolina
Con este simple ejemplo resulta evidente que el sitio de inicio de la traducción es de importancia capital para la correcta traducción del mensaje.
¿Qué determina que la lectura comience en el sitio correcto?
Los ARNm portan un codón, el AUG (codifica metionina), que es interpretado por los ribosomas como codón de iniciación. Este codón da el marco o cuadro de lectura que será empleado de allí en más. En adelante, el ribosoma se desplazará a lo largo del ARNm en bloques consecutivos de tres bases, garantizando la lectura de los codones apropiados.
Las mutaciones que implican ganancia o pérdida de un nucleótido resultan más destructivas que las sustituciones, pues al modificar el marco de lectura, alteran por completo el mensaje.
Por último hemos de señalar que el código es leído sin solapamiento. Esto significa que cada nucleótido del mensaje es utilizado como componente de un solo codón (aunque nuevamente existen excepciones).
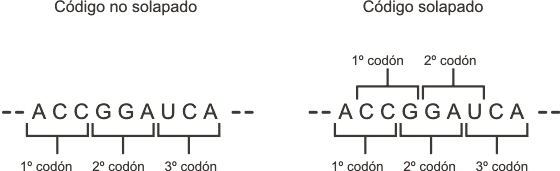
Fig. 11.2 - Solapamiento del código
Características del Código Genético
- El código genético consta de 64 codones o tripletes de bases.61 codones codifican aminoácidos.
- 3 codones funcionan como señales de terminación.
- El código no es ambiguo, pues cada codón especifica a un solo aminoácido.
- El código es degenerado, ya que un aminoácido puede estar codificado por diferentes codones.
- Es universal, debido a que sus mensajes son interpretados de la misma forma por todos los organismos.
- Utiliza un marco de lectura establecido al inicio de la traducción y no lo modifica.
- No se produce solapamiento de codones.
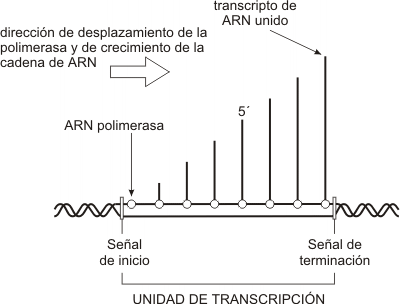
EL PROCESO DE LA TRANSCRIPCIÓN
La transcripción consiste en la síntesis de ARN a partir de un molde de ADN.
A continuación realizaremos una descripción general del proceso de transcripción tomando en cuenta los aspectos comunes a la síntesis de los distintos tipos de ARN.
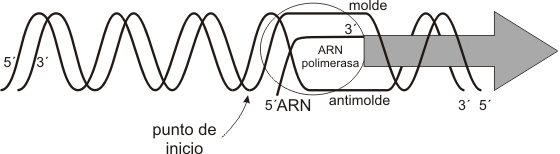
La transcripción, tanto en células procariotas como eucariotas, involucra la participación de una enzima ARN polimerasa ADN dependiente. Ésta sintetiza una cadena de ARN cuyos inicio, terminación y secuencia de bases vienen determinados por el propio gen.
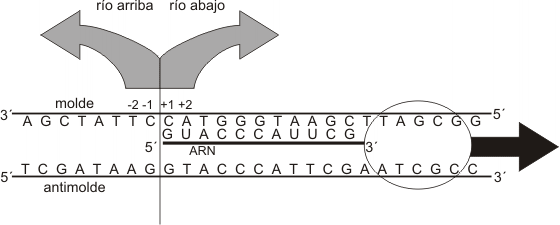
El primer paso de la transcripción es la unión de la enzima ARN polimerasa a una región del gen llamada promotor. El promotor es una secuencia específica de bases con alta afinidad por la enzima, por lo que proporciona a la misma su sitio de unión al ADN. Es asimismo una señal que indica cuál cadena se ha de transcribir. La transcripción es asimétrica, pues usualmente sólo se transcribe una de las dos cadenas que forman cada gen. La cadena que actúa como plantilla es la cadena molde, negativa o no codificante; la hebra no transcripta, complementaria de la anterior, se denomina antimolde, positiva o codificante. La ARN polimerasa se desplaza sobre la cadena molde, recorriéndola en dirección 3’ => 5’ o “río abajo” y transcribiéndola a partir del nucleótido que el promotor señala como punto de inicio de la transcripción. Este nucleótido se nombra +1 y los siguientes, río abajo, siguen la numeración correlativa (+2, +3, etc.). Partiendo desde el punto de inicio en la dirección contraria, es decir “río o corriente arriba”, los nucleótidos se numeran –1, -2, etc.
La ARN polimerasa sólo puede desplazarse y transcribir si previamente la doble hélice sufre un desenrollamiento y fusión (separación de las cadenas complementarias por ruptura de los puentes de hidrógeno entre las bases). La misma enzima cataliza ambos procesos, generando hacia el extremo 3´ una burbuja de transcripción, un tramo de aproximadamente 12 nucleótidos de longitud, en el cual las cadenas permanecen desapareadas. La burbuja de transcripción aparenta avanzar río abajo junto con la enzima, pues a medida que progresa la fusión por delante de ella, la doble hélice se recompone por detrás.
La formación de la burbuja causa una supertorsión o superenrollamiento de la doble hélice en los sectores ubicados hacia el extremo 5’ del molde. Este fenómeno es corregido por la acción de una enzima topoisomerasa I.
Cuando el molde ya ha sido desapareado en la burbuja de transcripción y expone sus bases, éstas son reconocidas por la ARN polimerasa. A medida que la enzima “lee” la plantilla, coloca junto a cada base de la misma el ribonucleótido trifosfato portador de la base complementaria. A los desoxirribonucleótidos de A, T, C y G se le aparean, respectivamente, los ribonucleótidos de U (recordemos que el ARN no contiene T), A, G y C mediante puentes de hidrógeno. Una vez ubicados los dos primeros ribonucleótidos, la enzima cataliza la formación del puente fosfodiéster entre ambos, iniciándose la cadena de ARN.
El enlace fosfodiéster se produce entre el hidroxilo en posición 3’ del primer nucleótido y el grupo fosfato interno, en posición 5’, del segundo. Un grupo pirofosfato de este último es liberado como producto y escindido rápidamente en dos fosfatos por la acción de una pirofosfatasa. Dicha ruptura es tan exergónica que la reacción inversa se torna prácticamente imposible. Así, desde el punto de vista termodinámico, se favorece la síntesis de la nueva cadena. Por lo tanto, son los mismos sustratos los que, por estar trifosfatados, aportan la energía necesaria para su polimerización.
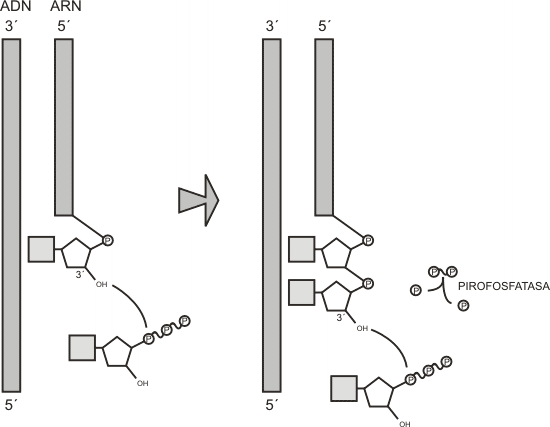
Este procedimiento se repite tantas veces como nucleótidos contenga el molde, de tal manera que el ARN va creciendo en forma antiparalela a aquél, es decir, desde su extremo 5’ (el que quedó libre en el primer nucleótido) hasta su extremo 3’ (que quedará libre en el último nucleótido añadido). La dirección de la síntesis del ARN es 5´ => 3´.
Siempre los nucleótidos recién incorporados al ARN forman una hélice corta ARN-ADN con su plantilla. Esta hélice híbrida es transitoria, pues conforme el polímero se alarga por su extremo 3’, el extremo 5’ se separa del molde, el cual vuelve a emparejarse con su hebra de ADN complementaria.
La transcripción concluye cuando la ARN polimerasa alcanza una señal (una secuencia específica de bases del ADN) que actúa como señal de terminación.
El producto obtenido, un ARN transcripto primario, resulta una copia complementaria y antiparalela de una región del gen comprendida entre el punto de inicio y la señal de terminación.
El transcripto primario repite la dirección y la secuencia (excepto porque lleva U en vez de T) de la hebra no copiada o antimolde, lo que justifica que a esta última se apliquen las denominaciones de hebra positiva o codificante. Es la cadena convencionalmente elegida para representar un gen.
Cabe aclarar que al describir la transcripción en forma general, hemos omitido la mención de regiones reguladoras de los genes, bajo cuyo control se encuentran los acontecimientos analizados. Este aspecto del tema será desarrollado más adelante en el mismo capítulo.
En el siguiente cuadro resumimos los requisitos de la transcripción:
|
· Una molécula de ADN molde, con región promotora, que indica el inicio de la transcripción, con secuencia de terminación, que marca el fin del proceso, y un segmento que será expresado. · Una enzima ARN polimerasa que: -reconoce las secuencias señalizadoras, -abre la hélice, -lee el molde (de 3´a 5´), -reconoce y ubica los sustratos complementarios, -polimeriza los sustratos(de 5´a 3´). · Cofactores enzimáticos de la ARN polimerasa: Mg++ o Mn++. · Sustratos: UTP, ATP, GTP Y CTP. · Una fuente de energía: los mismos sustratos. · Una pirofosfatasa. · Una topoisomerasa I. |
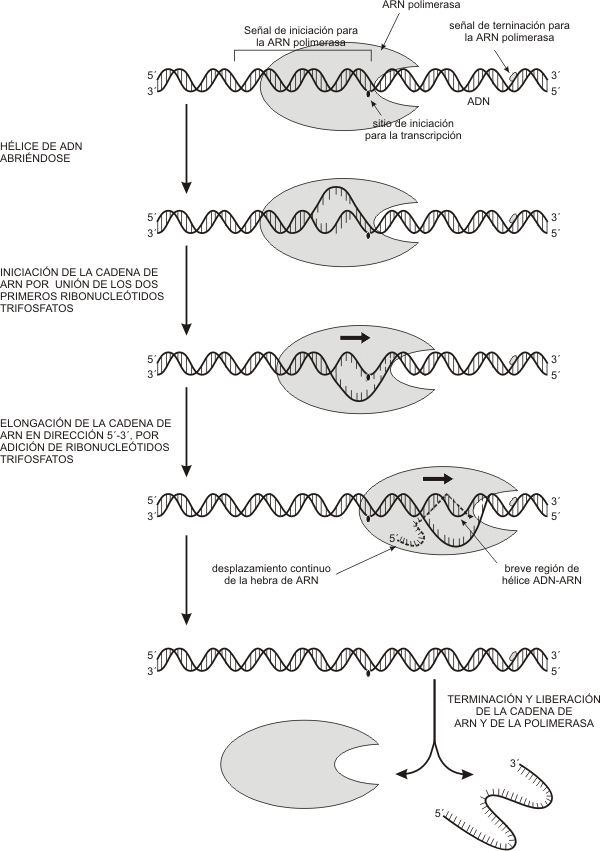
Fig. 11.7 - El proceso de transcripción
A partir de distintos genes se transcriben diferentes clases de ARN: ARN mensajero(ARNm),ARN de transferencia(ARNt),ARN ribosómico(ARNr),ARN pequeños nucleares y citoplasmáticos (ARNpn/ARNpc respectivamente).
Si bien el proceso transcripcional es básicamente similar en procariotas y eucariotas, existen diferencias que detallaremos a continuación:
|
EUCARIOTAS |
PROCARIOTAS |
|
1- La transcripción es llevada a cabo por tres tipos de enzima ARN polimerasa, cada una especializada en la síntesis de los distintos tipos de ARN: La ARN polimerasa I transcribe genes del ADN nucleolar que codifican para los ARNr 45S La ARN polimerasa II transcribe genes del ADN no nucleolar que codifican para todos los ARNm y para la mayoría de los ARNpn. La ARN polimerasa III transcribe genes del ADN no nucleolar que codifican para los ARNt, los ARNr 5S,los ARNpc y para el resto de los ARNpn. |
1- La transcripción es llevada a cabo por un solo tipo de ARN polimerasa que sintetiza las diversas clases de ARN. La ARN pol. bacteriana es un complejo proteico oligomérico constituido por cinco subunidades. Excepto la subunidad sigma (s), el resto conforma un núcleo enzimático. Todo el complejo constituye una holoenzima capacitada para leer las secuencias promotoras. |
|
2- Las ARN polimerasas eucariotas no se unen al ADN molde en su sitio promotor si no están presentes proteínas denominadas factores basales de transcripción. Estos son específicos de cada polimerasa y se encuentran en todos los tipos celulares. Las células eucariotas también cuentan con factores de transcripción específicos los cuales relacionan a los factores basales con las regiones reguladoras de un gen. Los factores específicos controlan la tasa de transcripción de los genes. Cada tejido presenta una combinación particular de los mismos. |
2- Si bien la ARN polimerasa bacteriana no requiere de factores de transcripción, se considera a la subunidad s como un factor de inicio de la transcripción, ya que el núcleo enzimático despojado de la subunidad s no puede por sí mismo leer adecuadamente las secuencias promotoras y efectuar la transcripción. |
| 3- Cada ARN polimerasa reconoce una secuencia particular de nucleótidos o
señal para la transcripción ,que es diferente para cada enzima. Por
ejemplo la ARN polimerasa II, que trasncribe los genes que codifican
para ARNm, reconoce varias secuencias de nucleótidos en el promotor,
entre las cuales se encuentran las secuencias llamadas caja TATA
o caja Hogness-Goldberg, caja CAAT y GC. Éstas secuencias
se encuentran "río arriba" respecto del punto de inicio
de la transcripción. El reconocimiento de dichas secuencias por parte
de la ARN polimerasa II y los factores basales de transcripción son
prerequisitos para iniciar la copia del gen.
Cabe destacar que las secuencias consenso que reconocen las distintas ARN polimerasas y sus correspondientes factores basales difieren en composición y ubicación dentro del gen, esto significa que no siempre se localizan delante del promotor. CAAT--GGGCCGGG--GGGCCGGG-TATA--inico Promotor eucariota
|
3- La ARN polimerasa bacteriana también reconoce secuencias consenso indispensables para la unión de la holoenzima y la señalización del punto de inicio. Una de las secuencias más conocidas es la caja TATAAT o caja Pribnow y TTGACA, ubicadas siempre río arriba del punto de inicio de la transcripción. -35 -10 +1 5' -----TTGACA----TATAAT----inicio--3' Promotor procariota
|
Resumiendo, las principales diferencias en la transcripción en células procariotas y eucariotas:
· Existe una sola ARN polimerasa procariota y tres ARN polimerasas eucariotas.
· La ARN polimerasa procariota no requiere factores de transcripción. Las eucariotas requieren la presencia de factores de transcripción basales y su actividad es regulada por factores de transcripción específicos.
· Las secuencias señalizadoras son diferentes.
LA MAQUINARIA TRADUCCIONAL
La traducción implica el funcionamiento coordinado de un gran número de moléculas entre las que se encuentran los distintos tipos de ARN. Una vez transcriptos cada ARN sufre una serie de modificaciones cuyas características difieren según hablemos de células procariotas y eucariotas. Describiremos cada ARN en general y luego sus variantes en ambos tipos celulares.
ARNm (mensajero)
Los ARNm son moléculas lineales de cadena simple en donde residen las instrucciones para la elaboración de un producto proteico.
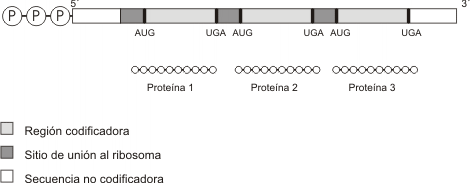
Los ARNm eucariotas y procariotas son esencialmente iguales en el sentido que presentan, una vez listos para la traducción, una secuencia continua de codones que se extienden desde el principio al fin del mensaje genético dictando con exactitud la secuencia lineal de aminoácidos de una cadena polipeptídica en particular. Esta secuencia se lee en la dirección 5' => 3'. El principio de la secuencia presenta un codón iniciador (casi siempre AUG), y el final de la secuencia o región codificadora es un codón de terminación o stop (UGA, UAA, UAG).
Además de la región codificadora presenta sectores extra en los extremos 5' y 3' del mensajero denominados secuencias directora y seguidora respectivamente. Estas regiones no se traducen en proteína aun cuando forman parte del ARNm transcripto del gen.
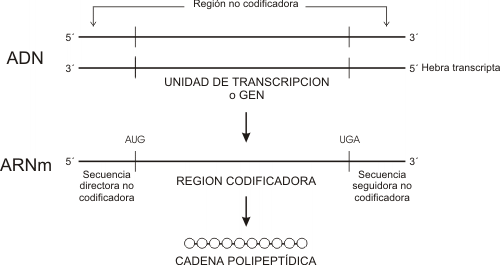
Existen diferencias entre los ARNm procariota y eucariota
ARNm eucariota
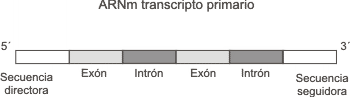
1- La mayoría de los ARNm eucariotas presentan la secuencia del mensaje interrumpida, es decir coexisten a lo largo de la molécula sectores que codifican para la proteína llamados EXONES, con secuencias intercaladas sin información denominadas INTRONES (Fig. 12.9).
2- Los ARNm transcriptos primarios sufren una serie de modificaciones antes de salir al citoplasma como ARNm maduro.
Algunos cambios consisten en el agregado de moléculas en los extremos 5' y 3' llamados capping y poliadenilación.
Capping: Ésta es la denominación del proceso por el cual se adiciona en el extremo 5' del ARNm una molécula de 7 metil-guanosina (un nucleótido metilado) a la que se conoce como cap (del inglés, capuchón). Ésta molécula se agrega al ARNm naciente cuando éste alcanza, aproximadamente, los 30 nucleótidos de longitud. Por ser esta modificación simultánea a la transcripción se la considera co-transcripcional. La cap impide la degradación del ARNm inmaduro por nucleasas y fosfatasas nucleares. También participa en la remoción de intrones y en el inicio de la traducción.
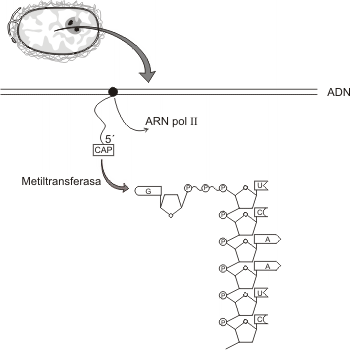
Fig 11.10- El agregado del cap
Poliadenilación: Este proceso consiste en el agregado de alrededor de unas 250 adenosinas
o cola poli A , en el extremo 3' del ARNm. El ARNm presenta una secuencia de
nucleótidos específica AAUAAA conocida como señal de poliadenilación. Una nucleasa corta
al pre-ARNm a unos 20 nucleótidos después de la señal. Una vez liberado el pre-ARNm, la enzima poliA-polimerasa le agrega las adenosinas de a una por vez. Por otra parte la ARN pol II continúa transcribiendo un tramo más del molde, para finalmente disociarse del gen. Este último tramo de ARN es totalmente infructuoso, pues resulta rápidamente degradado por nucleasas y fosfatasas.
Las funciones de la cola poli A son: proteger el extremo 3' de la degradación y ayudar a los ARNm a salir del núcleo.
Carecen de cola poli A los ARNm de histonas, dado que como ya se mencionó, no presentan la señal de poliadenilación. Otra excepción la ejemplifican algunos genes que presentan más de una señal de poliadenilación. Cuando así ocurre, el mismo gen puede ser transcripto en dos productos diferentes, dependiendo de cuál sea la señal reconocida.
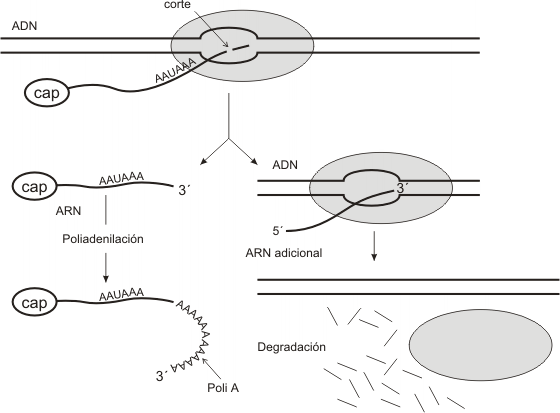
Otra modificación significativa afecta a la secuencia codificadora, que sufre un acortamiento producto de la eliminación de los intrones, quedando como producto final los exones empalmados en secuencia continua. Este tipo de procesamiento se llama empalme (splicing) y fue descubierto en 1977.
Para que se lleve a cabo este tipo de maduración del pre-ARNm se necesita una batería de ribonucleoproteínas nucleares: las RNPpn ( snRNP, del inglés, small nuclear ribonucleoprotein). Estas partículas ricas en uridinas y diversas proteínas se denominan U1, U2, U4, U5, U6 y se combinan de a una por vez en los extremos de cada intrón (lindantes a sendos exones). El complejo resultante del ensamblado de las distintas RPNpn se denomina espliceosoma. La actividad enzimática residiría en los ARNpn del espliceosoma. Éstos serían los responsables de reconocer las secuencias señalizadoras de corte, escindir a los intrones y empalmar a los exones entre ellos, produciendo una molécula de ARNm maduro.
Mecanismo molecular del splicing
El corte de intrones y el empalme de exones deben ser muy exacto, pues de lo contrario un error pequeño, causaría un corrimiento del marco de lectura del ARNm. Los intrones son clivados del transcripto primario por las RNPpn que reconocen cortas secuencias conservadas dentro del intrón y en los límites con los exones. Estas secuencias, muy similares en todos los intrones estudiados, son:
· Secuencia GU en el extremo 5' o sitio dador
· Secuencia AG en el extremo 3' del intrón o sitio aceptor
· Secuencias conocida como sitio de ramificación que se localiza en el interior del intrón
Estas secuencias participan en las reacciones de clivado y empalme que ocurren en dos etapas:
En la primer etapa, una RNPpn reconoce al sitio dador y otra se enlaza al sitio de ramificación. El extremo 5' es clivado y ligado a un nucleótido(A) del sitio de ramificación.
En la segunda etapa, se da el reconocimiento del extremo 3’ por parte de otra RNPpn y se produce el corte en el extremo 3' del intrón, seguido por el empalme de los dos exones. Así se libera el ARNm maduro del espliceosoma. El intrón queda eliminado en forma de lazo y será degradado posteriormente en el núcleo.
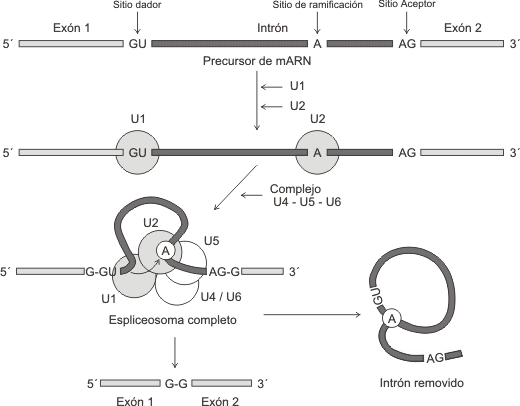
Como citáramos anteriormente, la presencia del capuchón en el extremo 5' es requerida para que las RNPpn trabajen, no así la cola poli A.
3- Los ARNm maduros son monocistrónicos, es decir el sector codificador dicta la secuencia para una sola cadena polipeptídica.
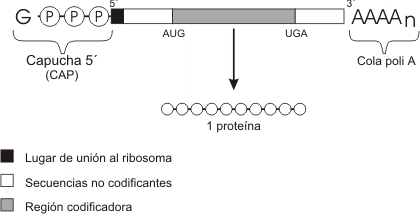
ARNm procariota
1-Los ARNm procariotas presentan las secuencias codificadoras continuas, pues carecen de intrones.
2-No sufren modificaciones post-transcripcionales.
3-Muchos ARNm procariotas son policistrónicos, es decir que una sola molécula de ARNm contiene información para varias proteínas. Este ARNm contiene codones para la terminación y para la iniciación de la traducción entre las secciones codificadoras de proteínas, de modo que se traduce como varias moléculas de proteína distintas.
ARNt (transferencia)
Los ARNt son moléculas "adaptadoras" ya que interactúan por un lado con la cadena polinucleotídica (ARNm) y por el otro con los aminoácidos que formarán parte de la cadena polipeptídica, así alinean a los aminoácidos siguiendo el orden de los codones del ARNm.
Cada ARN t es una molécula de 70 a 93 ribonucléotidos. Enlaces puente hidrógeno entre sus bases repliegan la molécula dando origen a una disposición espacial en forma de trébol (Fig.11.15). Cada brazo presenta una zona de doble cadena y un asa o bucle de cadena simple sin apareamiento de bases. Interacciones posteriores doblan la estructura de trébol formando una "ele".
¿Por qué existen 64 codones y aproximadamente 31 ARNt, si en la naturaleza hay 20 aminoácidos ?Existen 64 combinaciones posibles en que las 4 bases pueden ordenarse en tripletes o codones. De ellos, 3 son codones stop. Los 61 tipos restantes codifican para los 20 aminoácidos, existiendo para varios de ellos codones sinónimos (ver tabla 11.2).
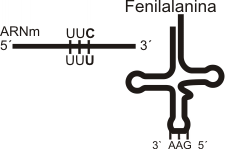
Los anticodones de los ARNt reconocen a los codones del ARNm, sin embargo no hay 61 tipos distintos de ARNt porque no se requiere la complementaridad exacta entre los 3 nucleótidos del codón y el anticodón.
En muchos casos, mientras coincidan las 2 primeras bases del codón, hay suficiente "balanceo"en la tercera posición para permitir el acoplamiento con más de un tipo de nucleótido en el anticodón, de manera que existen aproximadamente 31 tipos de ARNt.
Por ejemplo el aminoácido fenilalanina es codificado por dos codones sinónimo: UUU / UUC. Sin embargo un solo anticodón AAG puede complementarse indistintamente con UUU y UUC, realizando el trabajo de llevar a la fenilalanina al ribosoma para ser incorporada a la cadena polipeptídica en formación .
Todos los ARNt presentan un porcentaje significativo de bases poco frecuentes que surgen por modificación post-transcripcional de los cuatro ribonucleótidos estándar A, G, C y U (Fig. 11.16).
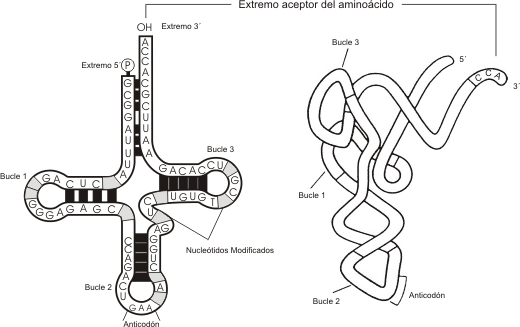
Fig. 11.15 - Estructura del ARNt
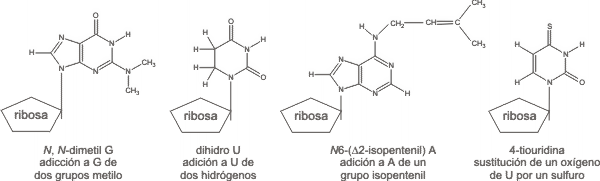
Fig. 11.16
- Bases raras en el ARNt
En esta molécula se distinguen básicamente dos extremos:
En el extremo 3' o extremo aceptor de la molécula se encuentra el trinucleótido CCA que representa el sitio de unión donde se liga el aminoácido. Esta unión es catalizada por una enzima aminoacil ARNt sintetasa específica y apropiada para cada uno de los 20 aminoácidos.
En el otro extremo de la "ele" se localiza un triplete de nucleótidos conocido como anticodón, cuya secuencia varía en cada tipo de ARNt. Cada anticodón es complementario de un codón del ARNm, aunque podría acoplarse a más de un codón (sinónimo).
Por lo hasta aquí expuesto, podemos concluir que el ARNt debe poseer varias propiedades específicas:
· Debe ser reconocido por una aminoacil ARNt sintetasa que lo una al aminoácido correcto.
· Debe tener una región que actúe como sitio de unión para el aminoácido.
· Debe tener una secuencia complementaria (anticodón) específica para el codón del ARNm correcto.
Las modificaciones que sufren los pre-ARNt son semejantes en procariotas y en eucariotas en cuanto en ambos se producen escisiones y modificaciones químicas, por ejemplo se elimina un dinucleótido 3' del precursor y se agrega el triplete CCA a los ARNt que no poseen todavía esta secuencia terminal. También aparecen bases raras (Fig.11.16) por modificación enzimática de nucleótidos estándar del ARNt precursor.
Algunos genes para ARNt presentan un intrón que interrumpe la secuencia codificadora, él cual es removido post-transcripción. (Fig.11.17)
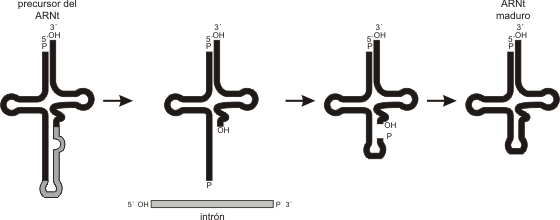
ARNr (ribosómico)
Los ARNr, junto a proteínas, son los componentes de los RIBOSOMAS. Los ribosomas y los ARNt intervienen en la traducción de la información codificada en el ARNm por lo cual los primeros son considerados fábricas de proteínas.
Cada ribosoma consta de dos subunidades: la subunidad MAYOR y la subunidad MENOR (Fig. 11.18)
La subunidad mayor contiene una depresión en una de sus superficies, en la cual se ajusta la subunidad menor. El ARNm se inserta en el surco formado entre las superficies de contacto de las subunidades. (Fig. 11.19).
Dentro de cada ribosoma existen tres huecos llamados sitios A (AMINOACÍDICO), P (PEPTIDÍLICO) y E (EXIT), para el ingreso de los ARNt unidos a sus correspondientes aminoácidos. (Fig. 11.20)
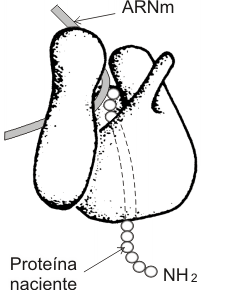
Fig. 11.19- Modelo de ribosoma que representa la ubicación tentativa del ARNm y de la proteína naciente
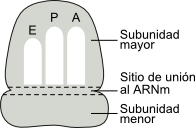
Fig. 11.20- Modelo esquemático del ribosoma y sus sitios A, P y E.
Las subunidades ribosómicas trabajan conjuntamente para la síntesis proteica. La subunidad menor aloja al ARNm sobre el cual se acomodan los ARNt para que puedan unirse los aminoácidos que transportan.
La subunidad mayor cataliza la unión peptídica de los aminoácidos gracias a la acción de la peptidil transferasa ( enzima que forma parte de la estructura de esta subunidad). [1]
Si bien cumplen idéntica función, los ribosomas procariotas y eucariotas se diferencian en su tamaño, coeficiente de sedimentación [2] , y en el tipo y número de moléculas de ARNr y proteínas que los forman:
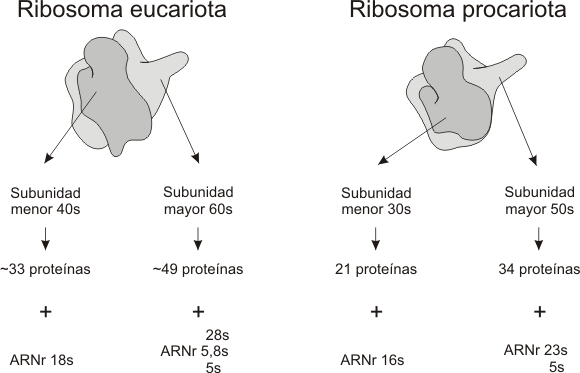
Fig. 11.21- Comparación de las estructuras de los ribosomas procariotas y eucariotas
Todos los ARNr procariotas y eucariotas sufren modificaciones post-transcripción.
En eucariotas los ARNr 18S; 5,8S y 28S son transcriptos de un mismo gen que codifica para un pre-ARNr 45S. La síntesis del este transcripto primario se realiza en la zona fibrilar del nucléolo a partir de la ARN pol I. Una vez obtenido el largo transcripto primario, se eliminan las secuencias espaciadoras(tramos de ARN inútiles para integrar la estructura ribosómica), las que serán degradadas por enzimas. Las secuencias resultantes utilizables de ARNr (28S, 18S y 5,8S) son metiladas y junto con ARNr 5S extranucleolar, se ensamblan a proteínas importadas del citoplasma para conformar la subunidades ribosómicas (Fig. 11.22).
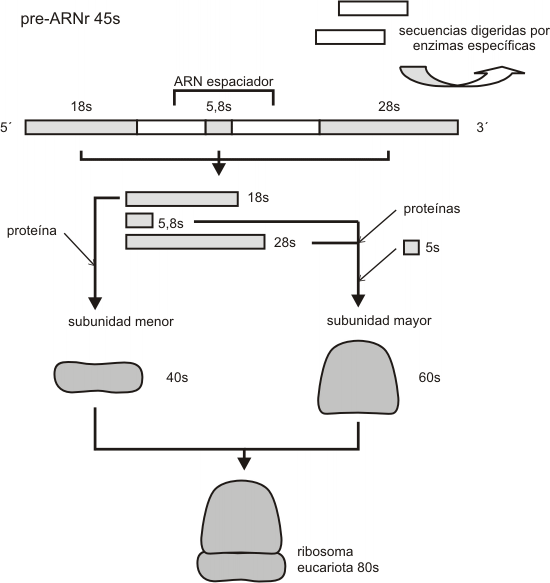
Fig. 11.22 - Procesamiento de los
ARN 45S
Las subunidades ribosómicas en distintos estadios de ensamblaje, se localizan en la periferia del nucléolo, conformando la zona granular del mismo.
Finalmente, las subunidades ribosómicas por separado y antes de completar sus respectivos ensambles, son exportadas al citoplasma a través del complejo del poro, concluyendo el procesamiento de cada subunidad en el citosol.
Respecto del ARNr 5S no transcripto en el nucléolo, una vez sintetizado se incorpora al resto de los componentes para conformar la subunidad mayor del ribosoma (Fig.11.23).
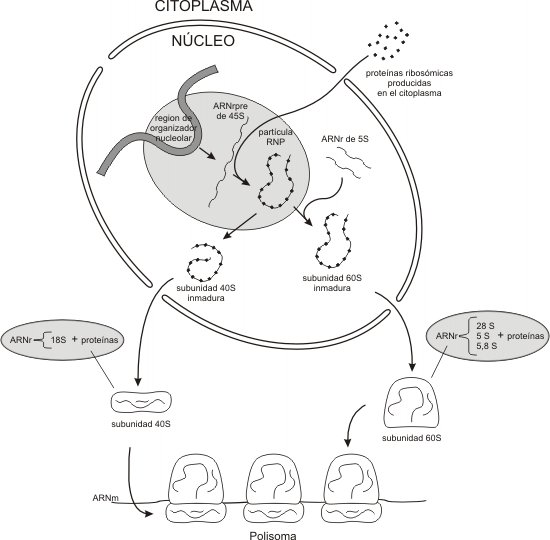
LOS ARN PEQUEÑOS
Los ARN pequeños forman complejos con proteínas específicas dando lugar a la formación de partículas ribonucleoproteicas (RNP).
Las RNP localizadas en el núcleo se denominan RNPpn: partícula ribonucleoproteica pequeña ( sn RNP/s: small -pequeña- /n: nuclear-RNP:ribonucleoproteica). Varios RNPpn conocidos como U1 a U6 participan en el procesamiento de los ARNm. Estas partículas forman un complejo multienzimático denominado espliceosoma, encargado de realizar cortes y empalmes en los ARNm transcritos primarios (splicing) .
Las RNP localizadas en citoplasma se denominan RNPpc: partícula ribonucleoproteica pequeña citoplasmática (scRNP/s: small/ c:cytosolic-citosol-/RNP:ribonucleoproteica). La función más conocida de cualquier RNPpc es la que desempeña el complejo ARN /proteínas que componen la partícula de reconocimiento de la señal o SRP. Estas partículas participan en el reconocimiento de secuencias específicas de las proteínas de secreción, membranares y lisosomales, deteniendo su traducción hasta que el ribosoma en donde se están sintetizando se contacte con la membrana del retículo endoplasmático granular, en donde finalizan su traducción.
EL PROCESO DE LA TRADUCCIÓN O SÍNTESIS PROTEICA
La síntesis proteica consiste en la traducción de la información codificada en la secuencia de nucleótidos del ARNm, en la secuencia correspondiente de aminoácidos en una cadena polipeptídica.
La síntesis proteica se lleva a cabo en los RIBOSOMAS y si bien en esencia es un proceso similar en todos los organismos, la maquinaria traduccional es más compleja en eucariotas.
La síntesis proteica incluye las siguientes etapas:
1. ActivaciÓn de los aminoÁcidos o aminoacilaciÓn
2. TraducciÓn del ARNm
1. ActivaciÓn de los aminoÁcidos
Antes de la traducción cada ARNt se engancha a su aminoácido específico. Esta reacción de aminoacilación es catalizada por las enzimas aminoacil ARNt sintetasas. Existen 20 tipos distintos de enzimas, una para cada aminoácido.
El proceso de aminoacilación ocurre en dos etapas:
a- En la primera fase se utiliza la energía de la hidrólisis del ATP para unir cada aminoácido a un AMP, con un enlace de alta energía. Esta reacción da origen a un complejo intermediario denominado aminoacil- AMP:
b- Sin abandonar la enzima, se transfiere el aminoácido del complejo aminoacil-AMP al ARNt específico, con lo cual se origina la molécula final: AMINOACIL -ARNt
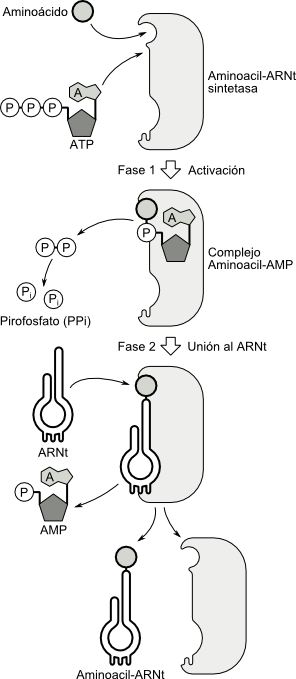
Fig. 11.24 - Etapas de la aminoacilación
En resumen, la aminoacilación o activación de los aminoácidos tiene dos funciones :
1- proporcionar el primer paso en la traducción del mensaje genético a una secuencia
de aminoácidos ;
2- activar al aminoácido antes de incorporarlo a la proteína. El enlace entre el ARNt y el aminoácido libera al hidrolizarse la energía necesaria para propulsar la formación del enlace peptídico durante la traducción.
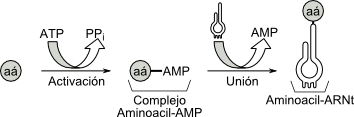
Fig. 11.25- Resumen del modelo de aminoacilación
2. Traducción
El mecanismo por el cual se traduce el mensaje del ARNm puede describirse en tres etapas:
· Iniciación
· Elongación
· Terminación
En todas las fases se requiere la presencia de un complejo sistema de proteínas citoplasmáticas conocidas como factores de iniciación, factores de elongación y factores de terminación respectivamente. Dichos factores son diferentes en procariotas y eucariotas aunque sus funciones son semejantes.
IniciaciÓn de la TRADUCCIÓN
En esta etapa se reúnen los componentes que constituyen el complejo de iniciación, disparador de la síntesis proteica. El complejo está compuesto por una molécula de ARNm, una subunidad mayor, una subunidad menor, el ARNt iniciador (cargado con N-formilmetionina en procariotas y con metionina en eucariotas) y factores proteicos de iniciación (IF). Dicho complejo comienza a formarse cuando se acoplan el ARNm y la subunidad menor del ribosoma. El ARNt iniciador ingresa al sitio P uniéndose por complementariedad de bases al codón AUG más próximo al extremo 5’ del ARNm. Este evento constituye una instancia crucial de la etapa de iniciación, pues garantiza que el mensaje genético se lea en sentido 5´=> 3´en el marco de lectura correcto para el resto de los codones que contenga la región codificadora del ARNm. La incorporación posterior de la subunidad mayor cierra el complejo de iniciación, originando un ribosoma completo y funcional.
Como todas las cadenas polipeptídicas son iniciadas por un ARNt iniciador, todas las proteínas recién sintetizadas tienen N-formil-metionina (o metionina en eucariotas) como residuo amino terminal. En ambos casos este aminoácido inicial puede ser eliminado por una aminopeptidasa específica, aunque a veces se mantiene en la proteína final.
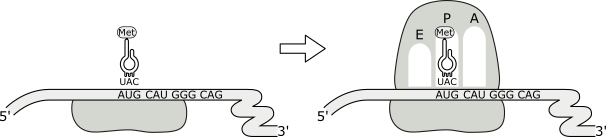
Fig. 11.26 - Fases de la etapa de iniciación de la síntesis proteica
En eucariotas, en cuanto se ha reconocido el codón AUG en el extremo 5' del mensaje, ningún otro codón AUG del ARNm será utilizado como lugar de iniciación, por lo tanto por cada molécula de ARNm se sintetiza una sola cadena proteica. Los ARNm eucariotas son monocistrónicos (Fig. 11.13 ).
En procariotas, dado que la secuencia de reconocimiento entre el ARNm y el ARNr de la subunidad menor puede aparecer varias veces a lo largo del mensaje, pueden originarse varias cadenas polipeptídicas a partir de un ARNm. La mayoría de los ARNm procariotas son policistrónicos (Fig. 11.14 ).
En conclusión, tres clases de interacciones determinan el inicio de la síntesis proteica:
- Reconocimiento del codón de iniciación AUG en la subunidad menor del ribosoma
- Acoplamiento de bases del codón AUG con el ARNt iniciador
- Acoplamiento de la subunidad mayor del ribosoma cerrando el complejo de iniciación
Las principales diferencias en la iniciación de la traducción en procariotas y eucariotas son:
|
Eucariotas |
Procariotas |
|
Modelo de selección: La subunidad menor se desliza sobre el ARNm hasta localizar al codón de iniciación. |
Modelo de emparejamiento: El acoplamiento de bases codón-anticodón iniciador se produciría por un emparejamiento de bases que preceden a AUG del ARNm con el extremo 3' del ARNr 16S de la subunidad menor. |
|
El ARNt iniciador transporta metionina. |
El ARNt iniciador transporta metionina formilada (N-formilmetionina). |
|
Se requiere la presencia de cap en el extremo 5'. |
No existe cap. |
|
La secuencia de iniciación aparece una vez a lo largo del mensaje (ARNm monocistrónico). |
La secuencia de iniciación puede aparecer varias veces a lo largo del mensaje(ARNm policistrónico). |
|
Participan factores de iniciación eIF (e= eucariota;I=iniciación;F=factor) específicos de este tipo celular |
Participan factores de iniciación IF (I=iniciación; F=factor) específicos de este tipo celular. |
ElongaciÓn de la cadena polipeptÍdica
El proceso de elongación o crecimiento de la proteína se inicia cuando una nueva molécula de aminoacil~ARNt ingresa al sitio A vacante del ribosoma (adyacente al sitio P que está ocupado) acoplándose por complementariedad de bases al segundo codón del ARNm expuesto en ese lugar. Esta reacción requiere la intervención de un factor de elongación y GTP.
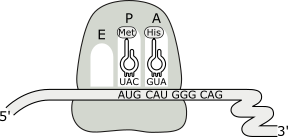
Fig. 11.27a- Ingreso del ARNt al sitio P durante la etapa de elongación.
El aminoácido iniciador se desacopla del ARNt del sitio P, liberando energía que se utiliza en la formación del enlace peptídico entre los dos aminoácidos alineados (reaccionan el carboxilo del primer aminoácido con el grupo amino del segundo aminoácido). Esta reacción es catalizada por una peptidil transferasa, ribozima integrante de la subunidad mayor. Como consecuencia de esta reacción, el ARNt iniciador del sitio P queda sin aminoácido y el dipéptido resultante queda enganchado al ARNt del sitio A (peptidil ARNt).
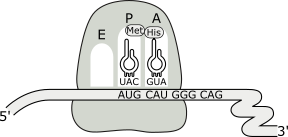
Fig. 11.27b - Formación de la unión peptídica durante la etapa de elongación.
El nuevo peptidil ARNt del lugar A es translocado al lugar P cuando el ribosoma se desplaza tres nucleótidos a lo largo de la molécula de ARNm. Esta etapa requiere energía y la presencia de un factor de elongación.
Como parte del proceso de translocación, la molécula libre de ARNt que se ha generado en el sitio P se libera del ribosoma, puesto que su lugar pasa a ser ocupado por el peptidil ARNt. El sitio de salida del ARNt descargado se denomina “E” (de “exit” o salida). Así el sitio A, que se halla desocupado, puede aceptar una nueva molécula de aminoacil~ARNt, con lo cual se vuelven a iniciar los pasos anteriormente analizados.
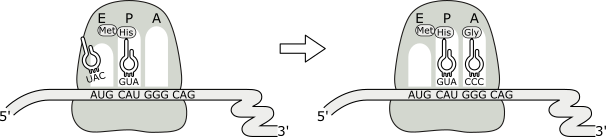
Fig. 11.27c - Translocación del ribosoma y elongación de la cadena peptídica.
Durante la elongación, como durante la aminoacilación, se ponen en marcha mecanismos correctores. En esta etapa se garantiza que el apareamiento de bases codón-anticodón sea correcto. Una equivocación en la correspondencia de nucleótidos daría como resultado la incorporación del aminoácido incorrecto. En este punto de control participa un factor de elongación que forma un complejo con el aminoacil ARNt y el GTP; este complejo y no el ARNt libre es el que se une al codón correspondiente en el ARNm. Recién después del correcto apareamiento codón-anticodón, el factor se disocia posibilitando la unión del aminoácido a la cadena polipeptídica. Este retraso en la unión del aminoácido posibilita que se elimine el ARNt incorrecto, antes que el residuo aminoacídico que transporta pueda enlazarse a la proteína en crecimiento.
Resumiendo, el ciclo de elongación de la síntesis se caracteriza por:
- La unión del aminoacil-ARNt (reconocido por el codón)
- La formación del enlace peptídico
- La translocación del ribosoma
TerminaciÓn de la síntesis proteica
La finalización de la síntesis proteica ocurre ante la llegada al sitio A del ribosoma, de uno de los tres codones stop o de terminación: UGA, UAG, UAA. Éstos son reconocidos por un factor de terminación o liberación. El factor de liberación se une al codón stop, estimulando la hidrólisis del enlace entre la cadena polipeptídica y el ARNt ubicado en el sitio P.
Como consecuencia de esta reacción el polipéptido se desacopla del ARNt, liberándose en el citoplasma. El ARNm se separa del ribosoma y se disocian las dos subunidades, las cuales podrán volverse a ensamblar sobre otra molécula de ARNm reiniciándose el proceso de traducción.
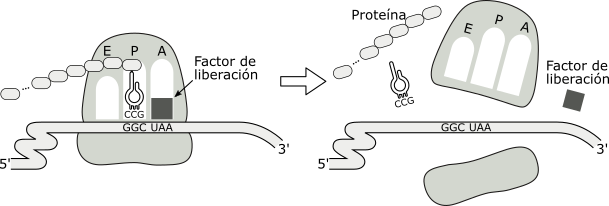
Fig. 11.28 - La etapa de terminación de la traducción
Los acontecimientos que caracterizan la terminación de la síntesis son:
- El reconocimiento del codón stop
- La disociación de las subunidades ribosómicas, el ARNm y la cadena polipeptídica.
El costo energÉtico de la síntesis proteica
La síntesis proteíca requiere más energía que cualquier otro proceso anabólico. Para formar cada enlace peptídico se consumen en total 1 ATP y 2 GTP, los que aportan cuatro enlaces de alta energía:- dos en la activación del aminoácido;
- el tercero en la unión del aminoacil ~ARNt a la subunidad menor del ribosoma;
- el cuarto en la translocación del ribosoma. Consideran éste cálculo, la síntesis de una cadena polipeptídica de 250 aminoácidos consume un total de 1.000 enlaces de alta energía. Si a esto sumamos el gasto que implica la transcripción de un ARNm que contenga como mínimo 250 codones, el costo energético se potencia aún más.
Polirribosomas:
Una molécula de ARNm dirige la síntesis simultánea de varias moléculas de una misma proteína. En cuanto un ribosoma ha traducido una secuencia de aminoácidos suficientemente larga como para dejar libre el extremo 5' del ARNm, un nuevo ribosoma puede iniciar la traducción. Al grupo de ribosomas que traducen simultáneamente el mismo mensaje se lo denomina polirribosoma o polisoma. Cada ribosoma en esta unidad estructural opera independientemente sintetizando una molécula de la misma cadena polipeptídica en distintos grados de crecimiento.
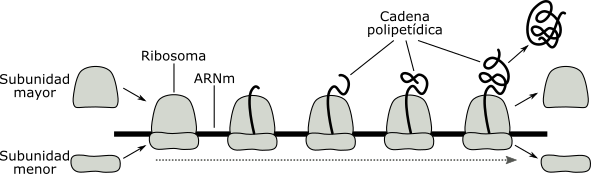
Fig. 11.29 - Esquema de un polirribosoma
Diferencias en la traducciÓn en procariotas y eucariotas
En eucariotas la envoltura nuclear y la maduración que sufren los ARNm impiden su traducción inmediata. El ARNm es "leído" después de que haya abandonado el núcleo a través de los poros nucleares. La traducción es por lo tanto post-transcripcional.
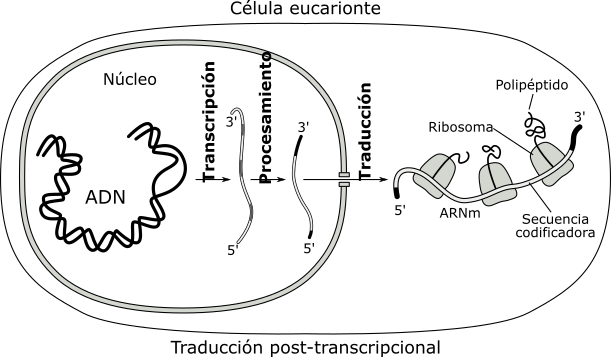
Fig. 11.30 - Transcripción, maduración y traducción en eucariotas

En procariotas la traducción es simultánea a la transcripción, esto es, mientras se está terminando de transcribir el extremo 3', el extremo 5' libre del ARNm se asocia a un ribosoma y al ARNt iniciador comenzando la traducción.
La fidelidad de la traducciÓn
La precisión en la incorporación de los aminoácidos en la secuencia apropiada durante la síntesis proteica, depende básicamente de dos mecanismos: La unión del aminoácido a su ARNt correspondiente o aminoacilación. En esta etapa la actividad correctora de las enzimas aminoacil - ARNt sintetasa minimizan los errores en la selección del aminoácido correcto. Muchas enzimas tienen dos sitios activos distintos, uno que lleva a cabo la reacción de carga, y otro que reconoce un aminoácido incorrecto que se haya colocado en el ARNt y lo libera por hidrólisis. El apareamiento de bases codón - anticodón, donde una equivocación en la correspondencia de nucleótidos daría como resultado la incorporación del aminoácido incorrecto. En este punto de control participa un factor de elongación que forma un complejo con el aminoacil ARNt y el GTP; este complejo y no el ARNt libre es el que se une al codón correspondiente en el ARNm. Recién después del correcto apareamiento codón - anticodón, el factor se disocia posibilitando la unión del aminoácido a la cadena polipeptídica. Este retraso en la unión del aminoácido posibilita que se elimine el ARNt incorrecto, antes que el residuo aminoacídico que transporta pueda enlazarse a la proteína en crecimiento.
REGULACIÓN DE LA EXPRESIÓN GENÉTICA
Regulación en procariotas: el operón
Las células procariotas pueden regular de varias formas la cantidad de proteínas que van a ser sintetizadas. No obstante se considera que la expresión génica está regulada principalmente a nivel de la transcripción .La mayoría de los procariotas, tales como Esclerichia coli, están expuestos a una gran variedad de condiciones en su medio y exhiben una notable capacidad para adaptarse a las mismas, esto es consecuencia de una gran habilidad para regular la expresión de genes específicos que codifican aquellas moléculas que responden a los estímulos de su entorno. Así, esta capacidad de un organismo para regular la expresión de sus genes incrementa su aptitud para crecer y dejar progenie en cualquier tipo de condiciones ambientales.La síntesis de transcriptos de genes y proteínas requiere un considerable gasto de energía. Si se "apaga" la expresión de estos genes (cuando sus productos no son necesarios). Un organismo puede evitar gastar energía o utilizarla en vías metabólicas de moléculas que maximicen la tasa de crecimiento en su medio circundante.¿Cuáles son los mecanismos por los cuales estos organismos regulan la expresión de genes en respuesta a cambios en el ambiente?En bacterias, los genes que codifican para la síntesis de enzimas que participan en una vía metabólica, se agrupan en el cromosoma en un complejo denominado OPERÓN. Todos los genes del operón actúan como unidades coordinadas mediante un mecanismo de control descripto por primera vez por Jacob y Monod en 1961.Un operón típico consta de: · Genes estructurales :son los genes que codifican para las enzimas de la vía metabólica. Tienen la particularidad de situarse próximos entre sí, de manera tal que son transcriptos en una sola molécula de ARNm policistrónico. Cuando éste se traduce se obtienen las diferentes enzimas de la vía metabólica. · Promotor: como analizáramos anteriormente, es la secuencia de nucleótidos del ADN en donde se une la ARN polimerasa para iniciar la transcripción. · Operador : es una secuencia de nucleótidos que se interpone entre el promotor y los genes estructurales, en donde se inserta una proteína reguladora denominada proteína represora.La proteína represora es codificada por el gen regulador, localizado en una región distinta del cromosoma bacteriano, aguas arriba del sitio operador.
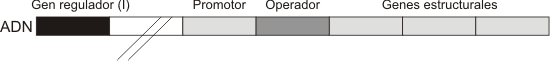
Uno de los ejemplos de operón más conocido es el operón lac. Este es un conjunto de genes que intervienen en la utilización de la lactosa, por parte de la bacteria, como fuente de energía. Las tres enzimas que intervienen en la vía de degradación de la lactosa son: la enzima permeasa , la beta galactosidadsa y la transacetilasa.El operón lac está formado por tres genes estructurales dispuestos en serie(z, y, a). La trasncripción de estos genes da origen a una molécula de ARNm que codifica para la tres enzimas que participan de la misma vía metabólica.En ausencia de lactosa el represor se enlaza al operador e impide a la ARN polimerasa insertarse en el sitio promotor con lo cual se interrumpe la transcripción (Fig. 11.33) .El represor ejerce su influencia mediante control negativo, puesto su interacción con el ADN inhibe la expresión del operón.
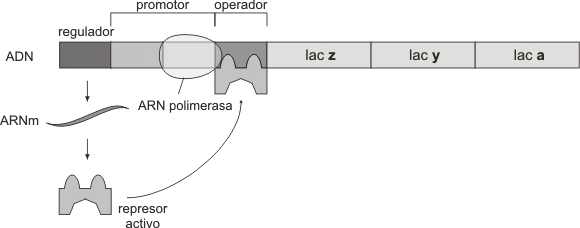
En presencia de lactosa, este disacárido se une a la proteína represora, provocándole un cambio conformacional e incapacitándola para unirse al ADN del operador. En estas condiciones, se transcriben los genes estructurales, apareciendo en el citosol las enzimas que degradarán a la lactosa. En este ejemplo de operón el represor solo puede unirse al operador en ausencia del inductor (Fig.11.34).
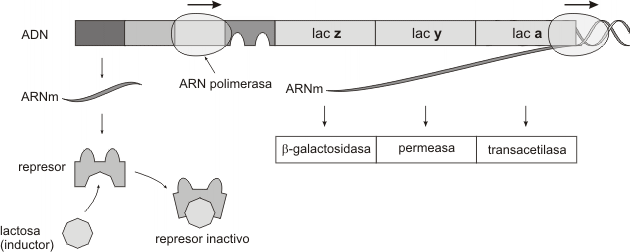
Fig. 11.34 - El operón lac activado
El operón lac es un ejemplo de operón inducible, es decir aquel en el cual la presencia de una sustancia específica (en este caso la lactosa) induce la transcripción de los genes estructurales.El operón lac también se encuentra bajo control positivo. Este efecto se da cuando en el medio hay glucosa, así la bacteria metaboliza este monosacárido ignorando cualquier otra fuente de carbono disponible. Cuanto menor es la concentración de glucosa en el medio, mayor es la concentración de AMPc , el cual tiene influencia en el "encendido" del operón lac.El AMPc actúa uniéndose a una proteína fijadora de AMPc denominada CAP (proteína activadora de catabolitos). Cuando la concentración de este complejo es alta (pues el medio contiene poca glucosa), el CAP-AMPc se fija a un sitio específico del promotor lac, aumentando la afinidad de la región promotora para la ARN polimerasa, lo que estimula la transcripción del operón (Fig. 11.35).
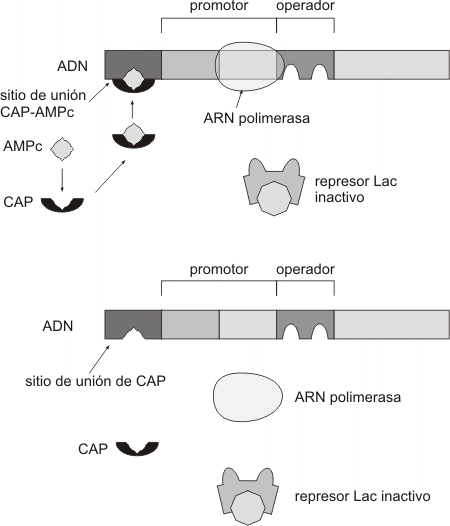
Por lo expuesto concluimos que:
Para que se exprese el operón lac deben darse dos condiciones en el medio: que esté presente la lactosa y que la concentración intracelular de glucosa sea baja.
Existen otros tipos de operones en bacterias, como por ejemplo el operón triptofano, el que se caracteriza por ser un operón reprimible.El operón triptofano consiste en cinco genes estructurales que codifican para las enzimas involucradas en la biosíntesis del aminoácido triptofano. Dichos genes se agrupan en una unidad de transcripción con un solo promotor y un operador. Un gen regulador se localiza fuera del operón y codifica para la síntesis de una proteína represora. Esta proteína difiere del represor lac en que se sintetiza en forma inactiva siendo incapaz de unirse al operador.En ausencia de triptófano, la ARN polimerasa se une al promotor y transcribe los genes estructurales en un ARNm policistrónico. Esto es posible pues el represor inactivo no logra unirse por si solo al operador (Fig. 11.36).
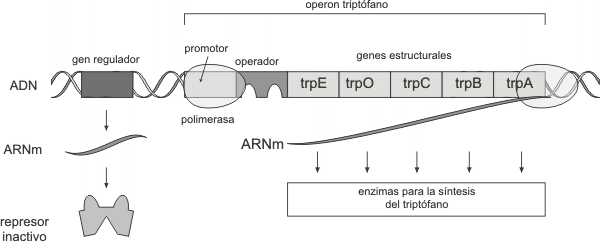
En presencia de triptófano en el medio circundante, el aminoácido (molécula denominada co-represor) se une a la proteína represora constituyendo el complejo represor/co-represor. Dicho complejo reconoce a la zona operadora a la que se fija, impidiendo a la ARN polimerasa transcribir los genes estructurales (Fig. 11.37).
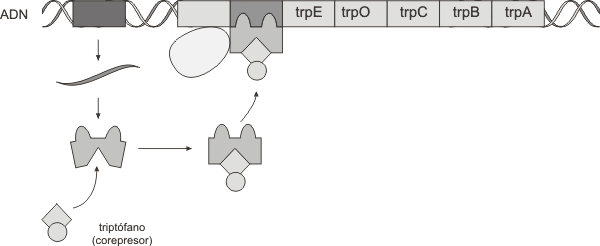
Con este mecanismo de regulación la bacteria ahorra energía sintetizando triptófano solamente cuando esta sustancia esencial en su crecimiento, está ausente en el medio ambiente.
Comparación entre el operón lac y el operón triptófano
|
OPERÓN LAC |
OPERÓN TRIPTÓFANO |
|
Operón inducible, se expresa en presencia de lactosa. |
Operón reprimible, se expresa en ausencia de triptófano. |
|
La lactosa es el inductor |
El triptófano es el co-represor |
|
El represor se sintetiza en forma activa. Actúa solo |
El represor se sintetiza en forma inactiva. Actúa en presencia del co-represor |
|
Sus enzima participan en un vía catabólica |
Sus enzima participan en una vía anabólica |
RegulaciÓn en eucariotas
Las células eucariotas presentan estrategias más complejas que las bacterias para regular la actividad de sus genes. Los organismos eucariotas pluricelulares poseen el mismo genoma en todas sus células, sin embargo los distintos tipos celulares se diferencian entre sí porque fabrican y acumulan distintos ARNm y por lo tanto distintas proteínas . ¿Por qué si el gen para la hemoglobina está presente en el genoma de una célula epitelial, no se encuentra esta proteína ni su ARNm en el citoplasma de este tipo celular?. ¿Cómo logran las células epiteliales mantener "apagado" el gen para la hemoglobina? .Para responder a estas preguntas debemos tener en cuenta no solo que el genoma eucariota es más complejo que el procariota, sino que existen más niveles en dónde ejercer el control de la expresión génica. Cada etapa en el flujo de información propuesto por el dogma de la biología molecular:
Si bien los mecanismos más importantes de control son los que actúan a nivel transcripcional, es importante destacar que pueden producirse regulaciones durante el procesamiento o maduración del ARNm o bien controlando: su pasaje a citoplasma o su supervivencia en el citosol. En algunos casos los controles actúan a nivel traduccional o regulando la actividad de la proteína.
Desarrollaremos
particularmente, aquellos mecanismos que operan a nivel transcripcional
es decir en el comienzo de la síntesis del ARNm ya que actúan sobre las
secuencias reguladoras del gen.
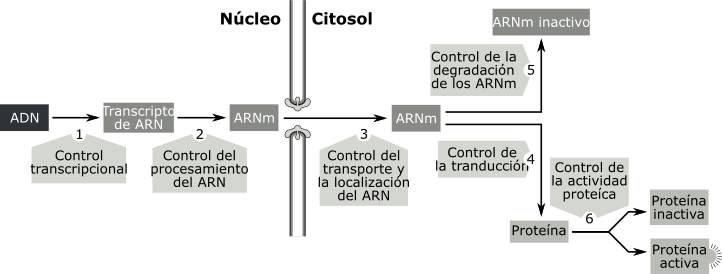
Fig. 11.38 - Niveles de control de la expresión génica
Control transcripcional
A) Los factores de transcripción y la expresión genética:Para la transcripción de un gen eucariota se requiere:
· Una secuencia promotora o promotor: secuencias de nucleótidos necesarias para la fijación de la ARN polimerasa.
· Secuencias reguladoras: existen dos tipos
Intensificadoras (del inglés, enhancers):secuencias que estimulan la transcripción y cuya localización puede ser a miles de nucleótidos de distancia "río arriba o abajo" del promotor.Silenciadoras (del inglés silencers) : secuencias que inhiben la transcripción. También pueden hallarse muy distantes del promotor.
· Factores basales de transcripción: complejo proteico que interacciona con el sitio promotor. Son esenciales para la transcripción pero no pueden aumentar o disminuir su ritmo. De esto se encargan: · Factores específicos de la transcripción: complejo de proteínas reguladoras que pueden ser activadoras o represoras.
Proteínas activadoras : interaccionan con las secuencias intensificadoras del gen.Proteínas represoras: interactúan con las secuencias silenciadoras del gen.
Estas proteínas reguladoras interactúan con regiones específicas del
surco mayor del ADN que corresponden a las zonas reguladoras del gen.
La geometría de la molécula, y los diseños característicos de los factores
de transcripción (Fig. 11.39), posibilitan dichas interacciones las que
se producen por uniones no covalentes del tipo puente hidrógeno, uniones
iónicas e hidrofóbicas.
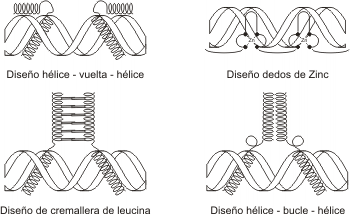 |
 |
Fig. 11.39 - Ejemplos
de diseños de factores de transcripción
Para comprender el mecanismo por el cual los factores de transcripción regulan la expresión de un gen eucariota, consideremos una analogía propuesta por Robert Tjian3 : " si el promotor fuese el encendido de un coche, los intensificadores serían el acelerador y los silenciadores los frenos, así las proteínas activadoras pisarían el acelerador y las represoras pisarían el freno". Entonces, la transcripción de un gen depende de la actividad conjunta de los factores de transcripción unidos a las secuencias reguladoras.
Cada gen tiene una combinación particular de intensificadores y silenciadores. Dos genes distintos pueden compartir idénticas secuencias intensificadoras y silenciadoras, pero no existen dos genes que posean la misma combinación de estas secuencias reguladoras.
Las diferencias entre los distintos tipos celulares que comparten el mismo genoma se deben a que cada célula transcribe y expresa distintas proteínas. Esto es consecuencia de que cada tipo celular contiene conjuntos de proteínas reguladoras de la expresión de diferentes genes.¿Cómo logran los activadores y silenciadores regular la transcripción si se encuentran a mucha distancia del promotor del gen ?La unión de los factores al sitio promotor provoca un cambio conformacional que pliega al ADN entre las secuencias reguladoras y promotora, formando un asa. Este plegamiento permite contactar a los factores específicos ,unidos a las regiones reguladoras con una o más proteínas "blanco" asociadas al complejo de transcripción basal. Cuando esto ocurre, se estimula la transcripción por parte de la ARN polimerasa la que se desplaza copiando la región codificadora del gen.Así como los factores de transcripción se asocian a las secuencias reguladoras, también se disocian. Dice Alberto Kornblihtt4: “Debilidad, reversibilidad y especificidad de unión son características primordiales de las interacciones entre moléculas para producir efectos biológicos. Una complicada red de interacciones entre macromoléculas, principalmente del tipo ADN-proteína y proteína-proteína, es la que enciende diferencialmente los genes en los distintos tipos celulares”.
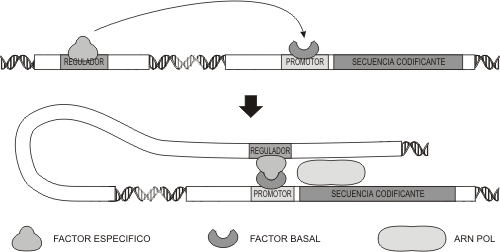
B) La estructura de la cromatina y la expresión genética
En cada tipo celular solo se expresan determinados conjuntos de genes mientras el resto del genoma se mantiene "silencioso". Se supone que el grado de compactación de la cromatina desempeña un importante papel en la expresión génica.Existen dos tipos de cromatina: la eucromatina, transcripcionalmente activa, más desplegada y la heterocromatina, transcripcionalmente inactiva, más condensada. La transcripción solo ocurre cuando el ADN está desplegado. Por lo tanto, las regiones de cromatina condensada y dispersa varían según el tipo celular, reflejando, según se cree, la síntesis de proteínas diferentes por distintos tipos celulares, es decir: el gen para la hemoglobina estaría en estado heterocromático en la célula epitelial y por lo tanto no disponible para su expresión.
C) El grado de metilación y la expresión genética
En algunos eucariotas la presencia de grupos metilo (CH3) en el gen afecta su expresión. La metilación ocurre en la citosinas que forman parte del dinucleótido -CG- dentro de secuencias específicas, con frecuencia localizadas dentro o próximas a la zona reguladora del gen. No todos los lugares CG están metilados. La mayoría de los genes que no se expresan están metilados, mientras que en otra célula en donde esos genes se expresan se advierte una disminución de sus niveles de metilación. Por ejemplo, en las células de mujeres, el cromosoma X condensado (el corpúsculo de Barr) presenta alto grado de metilación en los CG de los promotores de los genes que porta, inactivando así esta parte del genoma.
Control a nivel del procesamiento del ARNm
Se han descubierto formas de regulación que implican el procesamiento del ARNm. En algunos casos un mismo gen produce una proteína en un tejido y un tipo distinto de producto proteico en otro tejido.El mecanismo por el cual puede obtenerse de un mismo gen dos proteínas relacionadas se denomina empalme alternativo. Este proceso consiste en unir covalentemente diferentes combinaciones de exones del pre-ARNm obteniéndose dos ARNm maduros con distinta información y por lo tanto dos productos proteicos que difieren en uno o más tramos de su secuencia aminoacídica.
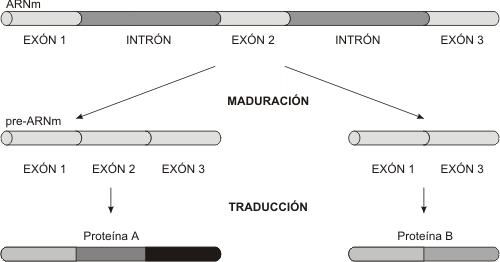
MECANISMOS DE CONTROL A NIVEL DE LA TRADUCCIÓN
Entre los ejemplos de mecanismos regulatorios de la traducción o síntesis proteica, explicaremos cómo se modifica la tasa de traducción del ARNm que codifica para la proteína ferritina. Esta proteína funciona capturando átomos de hierro del medio intracelular, ya que en estado libre, el hierro es tóxico para la célula.La traducción del ARNm para la ferritina es regulada por una proteína represora, la aconitasa, cuya actividad depende de la concentración de hierro libre en el citosol.Cuando la concentración intracelular de hierro es baja, la aconitasa se une a una secuencia nucleotídica específica en el ARNm, llamada elemento de respuesta al hierro (ERH), provocando un plegamiento del mensaje a modo de bucle, que bloquea la traducción (Fig.11.42).Cuando aumenta la concentración de hierro en el citosol, éste se asocia a la aconitasa, la cual cambia su conformación y pierde afinidad por el ERH. Una vez liberado el ARNm, comienza la síntesis de ferritina y su asociación con el hierro intracelular.Otro mecanismo importante es el control de la estabilidad del ARNm. Se conocen algunos casos en donde la integridad de la cola poli A es determinante para la supervivencia del mensaje. El acortamiento gradual de la cadena de adeninas por acción de nucleasas, reduce en algunos casos la vida media del ARNm.
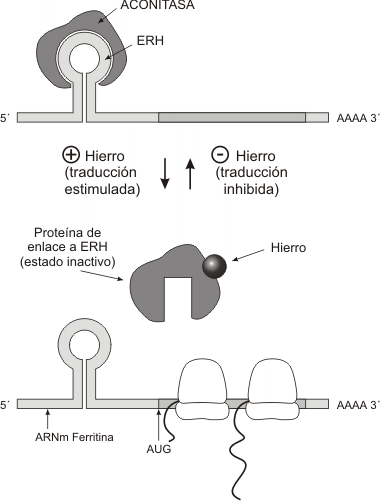
Fig. 11.42 - Control de la traducción del ARNm de la ferritina
MECANISMOS DE CONTROL DESPUÉS DE LA TRADUCCIÓN
La vida media de las proteínas puede ser considerada una forma indirecta de la expresión genética.Dos factores son determinantes de la vida media de una proteína citosólica: su correcto plegamiento y la secuencia aminoacídica de su extremo aminoterminal.Desde el momento que una proteína emerge del ribosoma citosólico, chaperonas moleculares de diversos tipos se unen a la cadena en síntesis, ayudándola a adquirir la conformación nativa. Esta unión transitoria evita que las proteínas recién sintetizadas alcancen espontáneamente un estado de agregación irreversible.Respecto del extremo aminoterminal, existen secuencias aminoacídicas estabilizadoras, que asegurarían una vida media prolongada y otras desestabilizadoras, las cuales favorecerían la marcación de las proteínas en dicho extremo. Tal marcación las conduciría a su degradación. Esto es conocido como la regla del aminoterminal.Si una proteína adquiere un plegamiento anómalo, o se ha desnaturalizado, o bien su extremo aminoterminal es desestabilizante, entonces ésta pasa a un proceso de ubiquitinización. La ubiquitinización consiste en la adición de varias moléculas de un polipéptido básico de 76 aminoácidos, muy conservado evolutivamente: la ubiquitina.La vía de la ubiquitina consta de varios pasos mediados por enzimas, que implican gasto de energía (ATP).
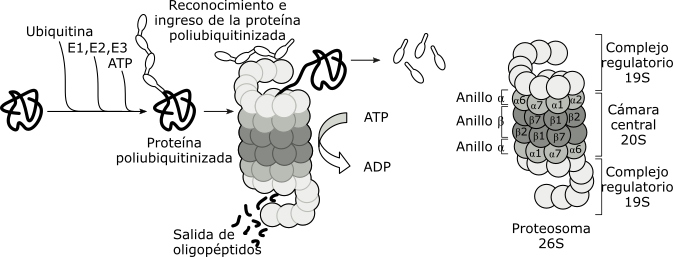
Fig. 11.43a - Ubiquitinización de proteínas mal plegadas y degradación en el proteosoma
Otra vía compite con la ubiquitinización. Las proteínas desnaturalizadas son conducidas por chaperonas moleculares hasta una chaperona oligomérica o chaperonina. Éstas pueden recuperar el plegamiento nativo de la proteína, en tanto se unan a ella en una etapa previa o temprana de la ubiquitinización. En caso contrario, la proteína ubiquitinizada, será captada por un complejo enzimático denominado proteasoma.
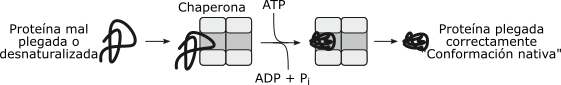
Fig. 11.43b - Acción de las chaperonas sobre las proteínas citosólicas
Los proteasomas están formados por más de una docena de proteasas, que
se organizan en cuatro anillos apilados que delimitan un cámara central.
En ambos extremos de este canal, se localizan sendos complejos regulatorios,
formados por diferentes ATPasas, y varios sitios de reconocimiento de
la ubiquitina.
La proteína ubiquitinizada es reconocida por la partícula regulatoria del proteasoma perdiendo su plegamiento por acción de las ATPasas, con gasto de energía. La proteína desenrollada es translocada dentro de la cámara central, donde las proteasas la degradan. Se producen oligopéptidos de aproximadamente 8 aminoácidos de longitud. La partícula regulatoria libera la ubiquitina para ser nuevamente utilizada. Tanto la actividad de las chaperonas como la de los proteasomas, aunque contrarias en su acción, garantizan la calidad de las proteínas presentes en el citosol.
Resumen de los mecanismos de regulación génica en eucariotas
|
Control transcripcional |
A- Factores de transcripciónB- Grado de condensación de la cromatina C- Grado de metilación |
|
Control procesamiento del ARNm |
Empalme alternativo |
|
Control transporte del ARNm |
Mecanismos que determinan si el ARNm maduro sale o no a citosol |
|
Control traduccional |
Mecanismos que determinan si el ARNm presente en el citosol es o no traducido |
|
Control de la degradación del ARNm |
Mecanismos que determinan la supervivencia del ARNm en el citosol |
|
Control de la actividad proteica |
Mecanismos que determinan la activación o desactivación de una proteína, como así también el tiempo de supervivencia de la misma. |
3 Robert Tjian enseña biología celular y molecular en la Universidad de California , en Berkeley, desde 1979.
4 Alberto Kornblihtt es Doctor en Química y Licenciado en Biología. Enseña Biología Molecular en la Facultad de Cs. Exactas y Naturales de la UBA.
BIBLIOGRAFIA
- Alberts, B et al. (2010). Biología Molecular de la Célula. 5ta. Edición. Ediciones Omega S.A. Barcelona.
- Becker W, Kleinsmith L y Hardin J (2007). El mundo de la célula. 6ta. Edición. Pearson Educación. Madrid.
- Cooper G, Hausman R (2010). La Célula. 5ta edición. Marbán Libros. Madrid.
- Curtis H, Schnek A, Massarini A. (2015). Invitación a la Biología en contexto social. 7ma. Edición. Editorial Médica
Panamericana. Bs. As.
- De Robertis(h), Hib J (2012). Biología Celular y Molecular de De Robertis. 16ma. Edición. Ediciones Promed. Bs.As.
- Karp, G. (2014). Biología Celular y Molecular. 6ta. Edición.Ed. Mc Graw Hill Interamericana. México
- Lodish H et al (2016). Biología Celular y Molecular. 7ma. Edición. Editorial Médica Panamericana. Bs. As.
- Pierce B.(2016). Genética. 5ta. Edición. Editorial Médica Panamericana. México.
[1] Recientemente se ha propuesto que algunos ARNr tienen actividad catalítica, con lo cual la función enzimática quedaría a cargo de un ARNr (ribozima) y no de la proteínas.
[2] Coeficiente de sedimentación o unidades Sverdberg (S) de sedimentación: mide la velocidad con que sedimentan los ribosomas u otras partículas o moléculas cuando se las ultracentrifuga.